《1. 引言》
1. 引言
合成生物学是生物学和工程学的结合,它试图通过应用工程学原理来设计细胞控制的遗传元件,以此获得对自然界的新认知和利用。合成生物学常见例子包括记忆开关[1]和振荡器[2]电路,它们可以使细胞根据化学或其他信号的输入,在不同的状态之间进行切换。这种感知环境输入的能力使细胞能够充当生物传感器。合成工程生物传感器[3–6]利用活细胞的多功能性,可以实现对输入信号的检测和对输出信号的分析。活细胞,尤其是细菌,可以在许多条件下存活[7,8],并且可以被设计成在临床环境中检测和诊断感染性的生物标志物。
转基因生物(genetically modified organism,GMO)受到诸多法律约束和政府机构的监管[9–13],这使得它们难以在实验室外进行应用。为了使工程生物传感器能够用于现场疾病诊断,必须满足相关规定。增材制造(俗称3D打印)采用三维打印的模式,它可以作为一种解决方案,使转基因生物在实验室外使用时与环境隔离。
30年前,核心增材制造方法首次实现商业化[14]。此后,增材制造技术发展迅速,3D打印机得到广泛应用。利用3D打印机连续沉积挤压出细丝层形成三维结构[15]可以完成该工作。此类3D打印机已经被证明能够成功地设计、打印和测试医疗相关设备[16,17]。
在这项研究中,我们选择了一种可以检测铜绿假单胞菌(Pseudomonas aeruginosa)的群体信号的稳定可靠方法,这是合成生物学中常用的方法[图1(a)]。活体生物传感器由工程化的大肠杆菌(Escherichia coli)组成,其被设计用来检测群体信号N-3-氧代十二烷酰-L-同型丝氨酸内酯(3O-C12)的存在。通过将铜绿假单胞菌的天然3O-C12信号通路的成分引入大肠杆菌,来构建活体生物传感器。在这里,转录因子LasR通过结合3O-C12和二聚体而充当信号转导子。然后,该复合物与同源启动子PLas [6,18,19]内的转录激活域结合,接着该转录激活域也被移植到大肠杆菌中。在我们的设计中,一旦结合,LasR就会促进红色荧光蛋白mCherry基因的表达。我们特别选择了3O-C12作为待检测的化学物质,因为它与医疗环境具有相关性[20–25]。
有一个关于该方法临床相关性的例子,在农村和欠发达地区,可以在当地诊所获得有机体铜绿假单胞菌的肺部感染病例。在铜绿假单胞菌感染肺部后[图1(b)],可在肺痰标本中检出该菌。该痰液可收集并置于市售样品管中。因此,通过利用合成生物学工具,能够在现场立即分析这些样品的方法将是早期诊断的理想选择。
《图1》
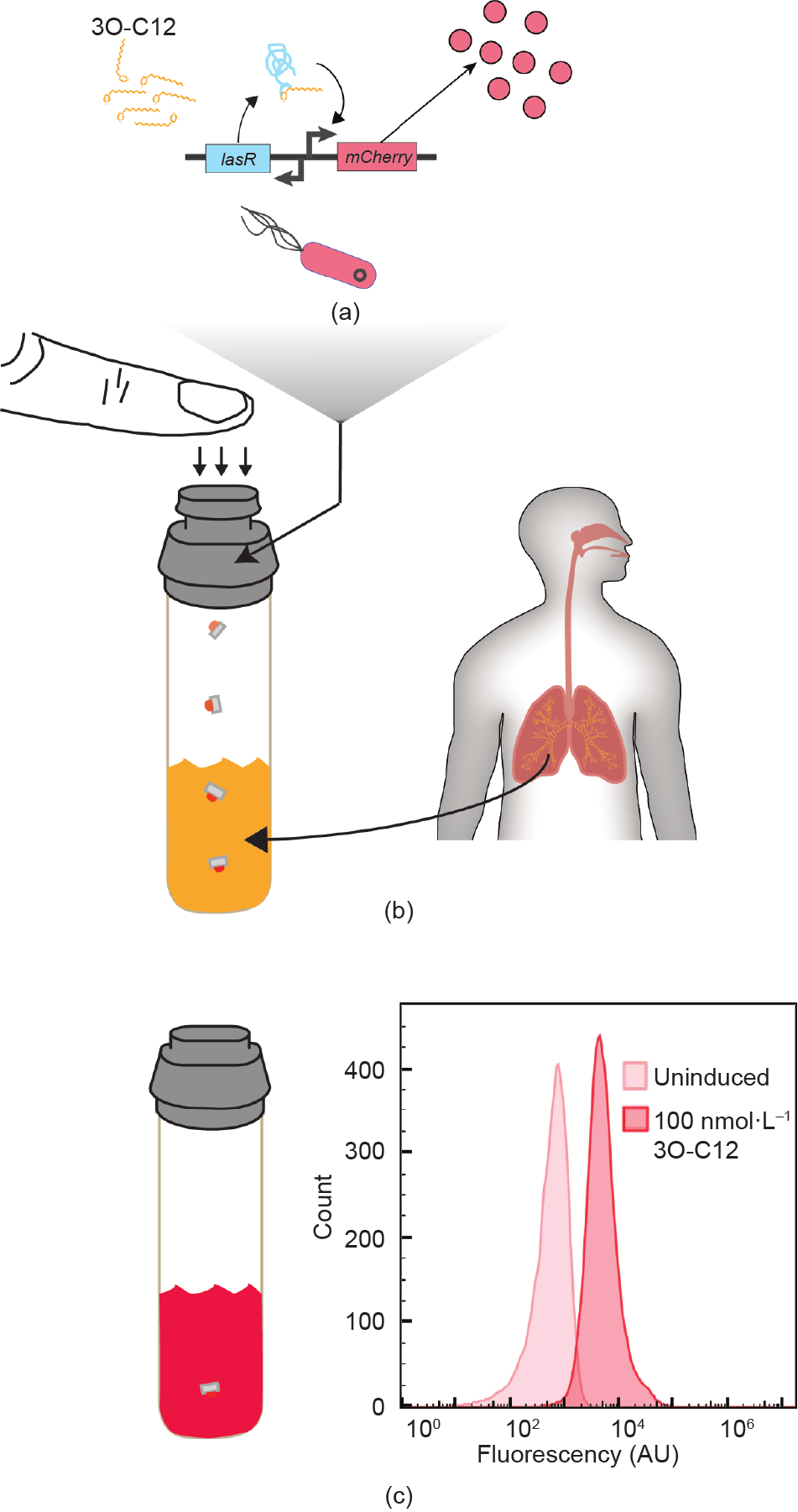
图1. 通 过 使 用GMO和3D打 印 试 管 盖 对3O-C12进 行 现 场 检 测。(a)3O-C12生物传感器能够持续表达一种可以与样品中的3O-C12分子结合的LasR蛋白。一旦与3O-C12结合,LasR蛋白二聚化并与PLas启动子中的DNA区域相结合,从而促进mCherry的表达。(b)将生物传感器放置在3D打印外壳中,提取生物样本并将其放置在连接到3D打印装置的样品管中,然后按下设备盖,使生物传感器落入样品中。(c)生物传感器变红,表明样品暴露于3O-C12,然后可以使用流式细胞术量化该信号。直方图表示针对未被诱导的和100 nmol·L−13O-C12样品(三个样品)的10 000个事件收集的数据。
在本文的解决方案中,合成生物传感器被置于一个可与普通样品管匹配的独特试管盖中[图1(b)]。这种独特的3D打印设备完全封装了活体生物传感器,允许其从实验室送出,同时还可以将传感器直接导入样本。当准备好分析痰液样本时,用手指向设备施加力,使细菌生物传感器从外壳中弹出并落入样品中。检测样品中由铜绿假单胞菌产生或已经存在于样品中的3O-C12,使活体生物传感器产生mCherry [图1(c)]并使样品变红,该颜色可通过荧光分析或肉眼来检测。因此,样本中的比色变化可以表明肺中是否存在铜绿假单胞菌。
铜绿假单胞菌是囊性纤维化患者的一种条件致病菌,它会引起肺部和呼吸道的反复感染。目前的诊断技术通常采用酶联免疫吸附测定(enzyme-linked immunosorbent assay,ELISA)[26]和实时聚合酶链反应(polymerase chain reaction,PCR)测定[27]的形式,这两种检测方法要么价格昂贵,要么需要大量的培训才能进行。本文所描述的传感方法可以作为当前检测铜绿假单胞菌诊断技术简单且廉价的补充。由于细胞生物传感器可以生长在一个安全的、3D打印的封闭设备内,因此该设备可以用作在现场部署合成工程生物传感器的平台。
《2. 材料与方法》
2. 材料与方法
《2.1. 细胞培养与分子克隆》
2.1. 细胞培养与分子克隆
本研究中使用的大肠杆菌菌株来源于大肠杆菌K12亲本菌株,见表1[28]。大肠杆菌细胞在37 ℃的溶菌肉汤(LB,赛默飞世尔科技公司,美国)培养基中分批培养[29]。使用100 μg·mL−1 卡那霉素(赛默飞世尔科技公司,美国)作为选择抗性转化剂。为了进行克隆,使用NEB® Turbo大肠杆菌(新英格兰生物实验室,美国)作为宿主来繁殖质粒。细胞在添加卡那霉素的37 ℃的LB培养基中生长。为了在平板上生长,LB培养基中添加2%琼脂(m/V)和卡那霉素。
本研究中使用的质粒列于表1,其通过标准分子克隆方法进行构建[29,30]。首先从质粒pKDT17扩增843bp的PLtetO-1-lasR DNA区构建传感器质粒,该质粒是从Addgene.org订购的,最初来自彼得 · 格林伯格(Peter Greenberg)博士的实验室。扩增711 bp的mCherry基因产生一个RBS-mCherry-t0片段,将其插入pKDT17以获得LasR传感器质粒pDW065(图2)。
《表1》
表1 质粒、菌株、相关特征及来源一览表

《图2》
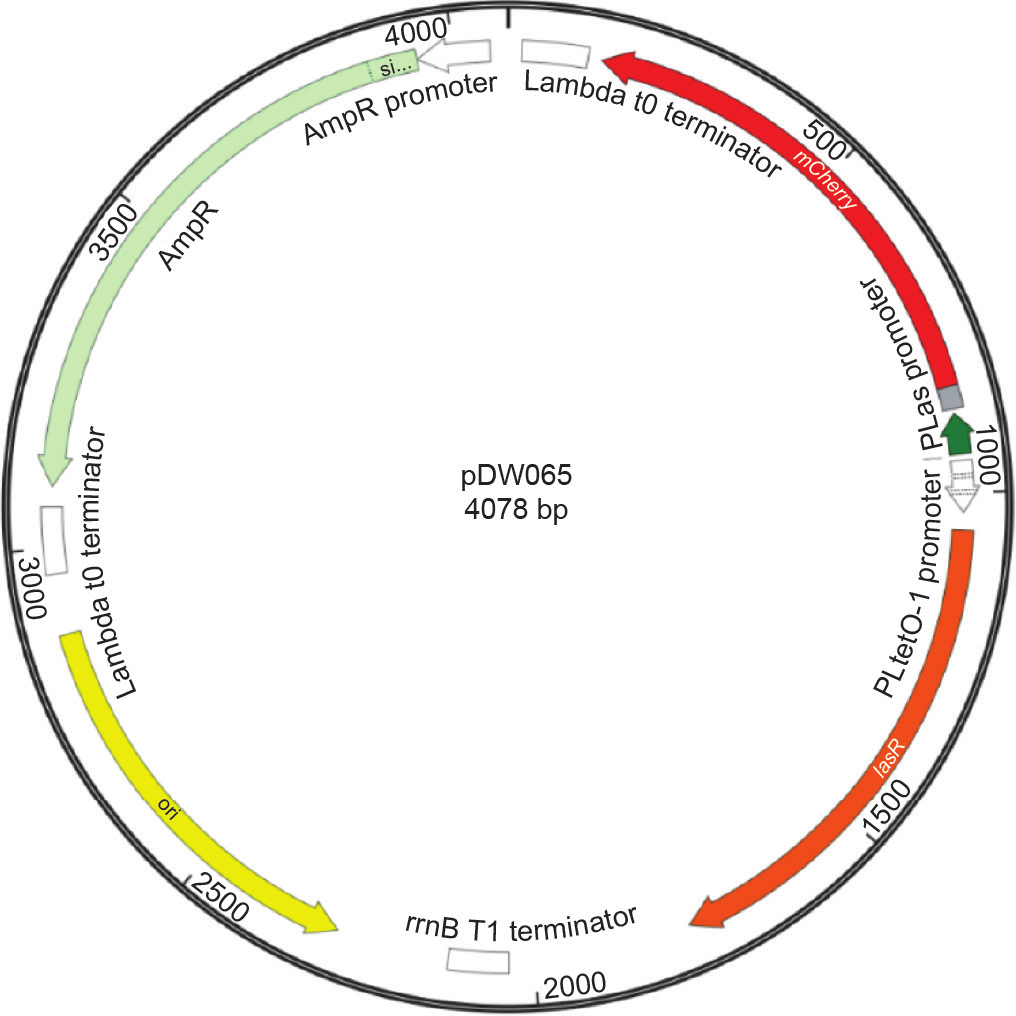
图2. 3O-C12传感器质粒图谱。3O-C12传感器也称为pDW065,由三个主要元件组成:质粒骨架、lasR片段和mCherry片段。骨架由氨苄青霉素抗性盒和pUC复制起点组成。将lasR基因置于PLtetO-1启动子的控制下,而mCherry基因则处于PLas启动子的控制下,该启动子含有LasR蛋白的结合位点。
《2.2. 剂量反应测量》
2.2. 剂量反应测量
为 了 研 究 该 基 因 网 络 的 剂 量 反 应, 在 添 加50 μg·mL−1 卡那霉素的LB培养基中,将具有pDW065的大肠杆菌的起始培养物培养过夜。将培养物稀释至0.2 OD600 (即光密度为600 nm),并使其生长直至达到0.6 OD600 。然后再次稀释培养物,补充0~100 μmol·L−1 3O-C12(西格玛-奥尔德里奇公司,美国),并在37 ℃下维持指数期直至完全诱导(约8 h)。将0.6 OD600 的培养物在过滤过的磷酸盐缓冲盐水(phosphate-buffered saline,PBS,赛默飞世尔科技公司,美国)中稀释至1∶100,并使用AccuriTM C6流式细胞分析仪(BD公司,美国)测量荧光。
《2.3. 丙烯腈 - 丁二烯 - 苯乙烯设备的设计与制造》
2.3. 丙烯腈 - 丁二烯 - 苯乙烯设备的设计与制造
3D打印设备采用Autodesk Inventor® (欧特克公司,美国)进行设计,并进行渲染成3D模型。然后将设计的模型导出为STL文件,该文件格式可支持Z-suite®(Zortrax公司,波兰)软件打开,该软件可将3D对象转换为一组打印机指令。然后,使用Z-suite® 软件将文件导出为z-code文件,该文件用于Zortrax M200(Zortrax公司,波兰)中,以使用丙烯腈-丁二烯-苯乙烯(acrylonitrile butadiene styrene,ABS)变体ABS ULTRAT打印3D设计。该专利ABS变体由ABS(90%~100%)、稳定剂(0%~5%)、润滑剂(0%~2%)、矿物油(0%~4%)、牛油(0%~4%)、蜡(0%~4%),聚碳酸酯(PC,0%~3%)和抗氧化剂(<2%)组成。
《2.4. 设备表面抛光》
2.4. 设备表面抛光
为了使打印后设备组件的表面粗糙度平滑,首先将3D打印组件放置在无菌表面上。将纸巾附着于1000 mL烧杯的内表面,并用丙酮浸透。将烧杯倒扣于设备部件上,并注意避免液体丙酮和部件之间的直接接触。1 h后,移除烧杯,将设备暴露于环境空气中2 h。
《2.5. 设备使用与操作》
2.5. 设备使用与操作
为了将3O-C12活体生物传感器包裹在3D打印装置中,首先在外壳的细菌孔中添加40 μL的琼脂塞,然后将工程化的大肠杆菌细胞接种到琼脂表面上。在过夜培养后,将柱塞压入外壳中并形成压合密封,借此将细菌细胞与环境隔离。然后,可以将包含活体生物传感器的盖子安全地转移到现场,并在使用前储存在冰箱中长达30 d。准备使用时,将待检样品放入14 mL的培养管中。移除培养管帽,将含有活体生物传感器的组装设备咬合到培养管上。为了激活生物传感,在紧握试管的同时按压盖子,直到细菌孔落入培养管中。然后将细胞培养物在37 ℃下培养8~9 h,随后用荧光光谱法或简单地用人眼检测。对于本研究中使用的流式细胞术分析,在OD600 为0.6时采集样品,并在去离子水中稀释至1∶100。使用AccuriTM C6流式细胞分析仪获取荧光测量值。实验完成后,可以在高压灭菌锅中对系统进行消毒;或者,如果没有高压灭菌锅,则可以拆卸部件并将其浸入次氯酸钠(即漂白剂)中。
《2.6. 基因调控网络建模》
2.6. 基因调控网络建模
我们设计了一种大肠杆菌生物传感器,当暴露于3O-C12时能够表达mCherry。活化剂LasR是在非活性状态下生成的。当将3O-C12引入细胞时,它与LasR相结合。该复合物激活基因调控网络。由于我们没有设计任何转录后调控,调控基因网络的一般行为可以通过转录反应位点的动力学建模。
为了捕捉系统的潜在动态,我们决定采用米氏方程来进行建模转录。当使用高拷贝数质粒时,我们可以假设随机效应对于特定的遗传组分是可以忽略不计的。这允许我们用一组常微分方程来简化模拟。
假设诱导物3O-C12在反应室内经过充分混合,且3O-C12在细胞溶质中的转运在单个细胞之间是一致的,我们可以模拟对3O-C12的转录反应,如式(1)所示。值得注意的是,该等式假定大多数转录后事件(翻译和蛋白质成熟)在对mRNA产生的反应一致且线性地发生。如果还假设核糖体结合位点强度是恒定的,并且细胞内存在丰富的资源和核糖体,那么这便是一个合理的假设。
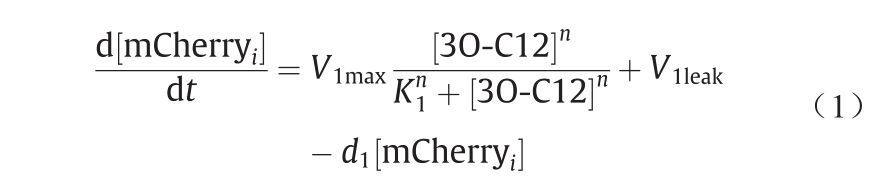
式(1)涉及mCherry的时间变化率与驱动转录事件的许多输入和参数的关系。
该模型固有的假设是细胞内的LasR浓度保持相对恒定。式(1)右侧的第一项为Hill函数[31],它将细胞内3O-C12的浓度与mCherry产生的速率相关联。这一项是阿拉伯糖浓度的函数,但它包括最大转录率 和动力学系数
和动力学系数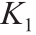 的参数。此外,Hill系数n与RNA聚合酶和启动子位点结合的协同程度有关。
的参数。此外,Hill系数n与RNA聚合酶和启动子位点结合的协同程度有关。
等式(1)右侧的第二项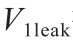 描述了启动子位点的“泄漏”。在物理条件下,这是在没有3O-C12的情况下产生的mCherry的速率。右侧的最后一项描述了细胞内mCherry的降解速率。该术语将衰变常数
描述了启动子位点的“泄漏”。在物理条件下,这是在没有3O-C12的情况下产生的mCherry的速率。右侧的最后一项描述了细胞内mCherry的降解速率。该术语将衰变常数 与mCherry的浓度[mCherryi ]相关联。为了更好地预测和建模基础动力学,这个方程可能适合于实验数据。
与mCherry的浓度[mCherryi ]相关联。为了更好地预测和建模基础动力学,这个方程可能适合于实验数据。
《2.7. 模型推导——剂量 - 响应曲线拟合、模拟和绘图》
2.7. 模型推导——剂量 - 响应曲线拟合、模拟和绘图
根据式(1),使用MATLAB ® (迈斯沃克公司,美国)优化工具箱分析和拟合实验数据。一旦确定了参数值,就可以使用MATLAB曲线拟合工具箱确认拟合。使用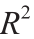 值评估模拟方程的拟合优度。仿真是用Python编写的,并使用FORTRAN库中的LSODE [32]进行数值集成。使用Matplotlib库在Python中绘制包含有实验数据的模拟。
值评估模拟方程的拟合优度。仿真是用Python编写的,并使用FORTRAN库中的LSODE [32]进行数值集成。使用Matplotlib库在Python中绘制包含有实验数据的模拟。
《2.8. 模型推导——生物传感器的有限元分析》
2.8. 模型推导——生物传感器的有限元分析
使用COMSOL(COMSOL Multiphysics® v5.2,COMSOL集团,瑞典)进行有限元分析。所有的模拟结果均代表了使用固体力学模块执行的静态研究。将Autodesk Inventor实体模型文件导入COMSOL中,以定义这些模拟的几何图形。然后将材料特性输入COMSOL以定义ABS材料的特性,从而便于进行分析。最后定义边界约束和应用载荷。此处显示的所有仿真图均由使用COMSOL的集成图形生成。
《3. 结果》
3. 结果
为方便使用,本文所述的3D打印检测装置可用于普通的细菌培养试管上[图3(a)],以确保使用方便。该设备由两个部件[图3(b)]组成,在此称为“外壳”和“盖子”。盖子用于将封闭在外壳内部的内容物与周围环境隔离。此外,盖子的顶部设计符合人体工程学,可用拇指或食指按压。组装完成后,可将设备封装在尺寸约为0.026 m×0.026 m×0.023 m的矩形棱柱内。组装完成的设备的横截面示意图[图3(c)]显示了通过压合连接在一起的外壳和盖子。外壳内有一个细菌孔,里面含有营养丰富的琼脂和一层细菌菌落,这用于维持和容纳设备内的细菌生物传感器。接下来,我们为设备增加了一些小型机械特征,其设计基于利用有限元方法软件套件[33]COMSOL Multiphysics® 进行建模。值得注意的是,我们的模型基于ABS(一种热塑性塑料)的结构。热塑性聚合物在加热后变得柔韧,在冷却时硬化,常用于3D打印。由于ABS的玻璃化转变温度为105 ℃,因此其非常适合3D打印,因为许多3D打印机的挤出温度设定为230 ℃ [34]。这种高打印温度也提高了打印设备的无菌性。
《图3》
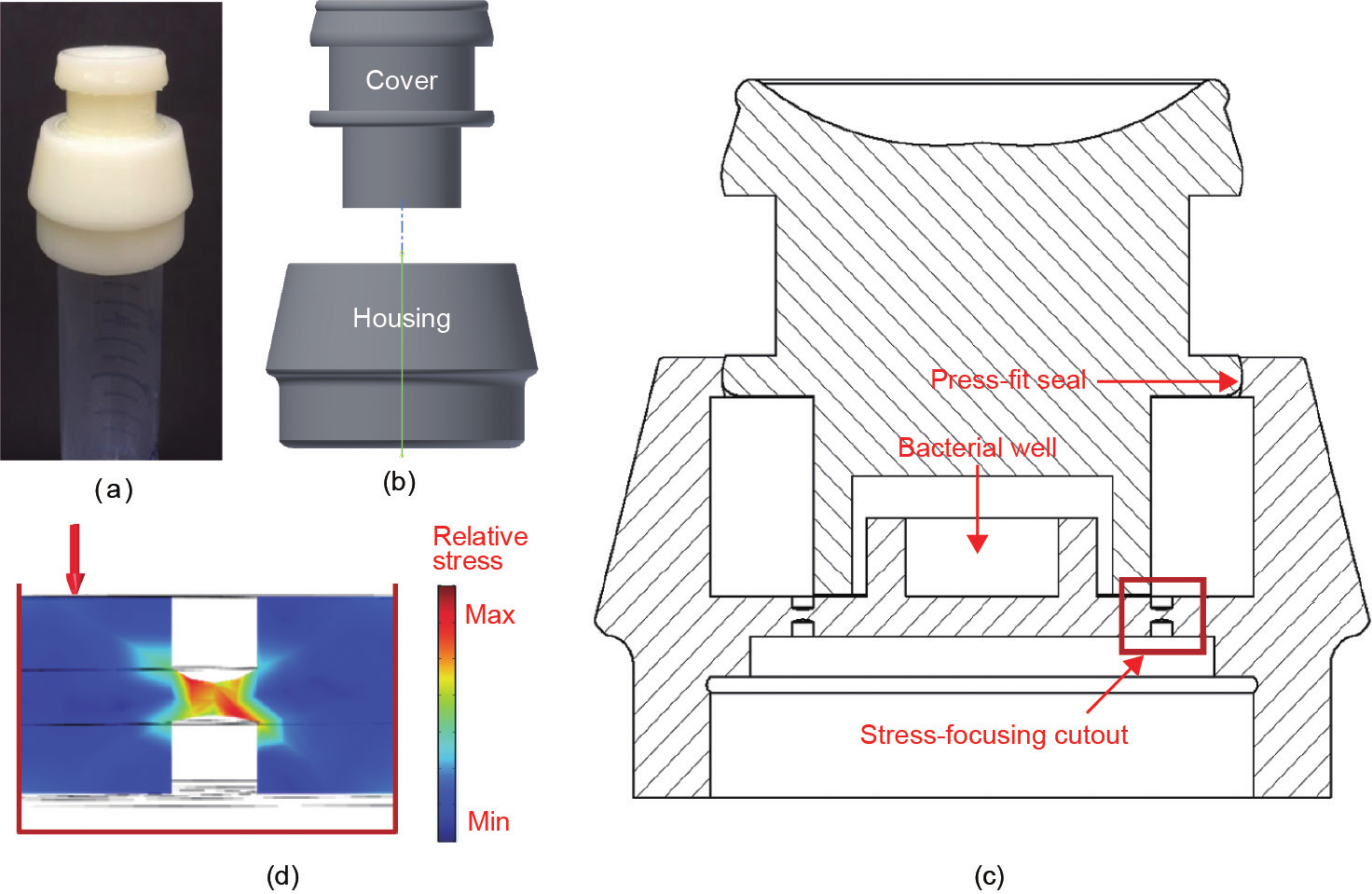
图3. 设备细节和原理图。 (a)3D打印设备可以连接到试管上,在试管中形成密封,该装置分为两部分:盖子和外壳; (b)设备盖压合密封在外壳上;(c)设备外壳包含细菌培养孔,作为沉积培养营养物、琼脂和活体生物传感器的腔室,壳体设计有应力聚焦切口,用于集中从盖子传递的压力,以便将细菌孔与外壳分离;(d)通过应力聚焦切口集中的应力使用COMSOL建模,以确保该装置在单手横向捏握时可用。Max:最大值;Min:最小值。
除了上面讨论的盖子、外壳和细菌孔特征外,我们还设计了一个小的机械特征,使轴向力(即沿着样品管的主轴直接向下施加在盖子上的力)引起外壳的底部快速地与外壳分离,从而在(设计的)灾难性故障期间将生物传感器释放到样品中。为了实现这种快速弯曲,我们在外壳底部的内部和外部创建了两个凹陷的环形特征,如图4和图5所示。在这里,我们将这些环形称为“应力聚焦切口”。这些切口特征用于集中施加在盖子上的轴向力所产生的应力,从而允许使用夹持器通过用户的拇指简单地激活设备。
《图4》
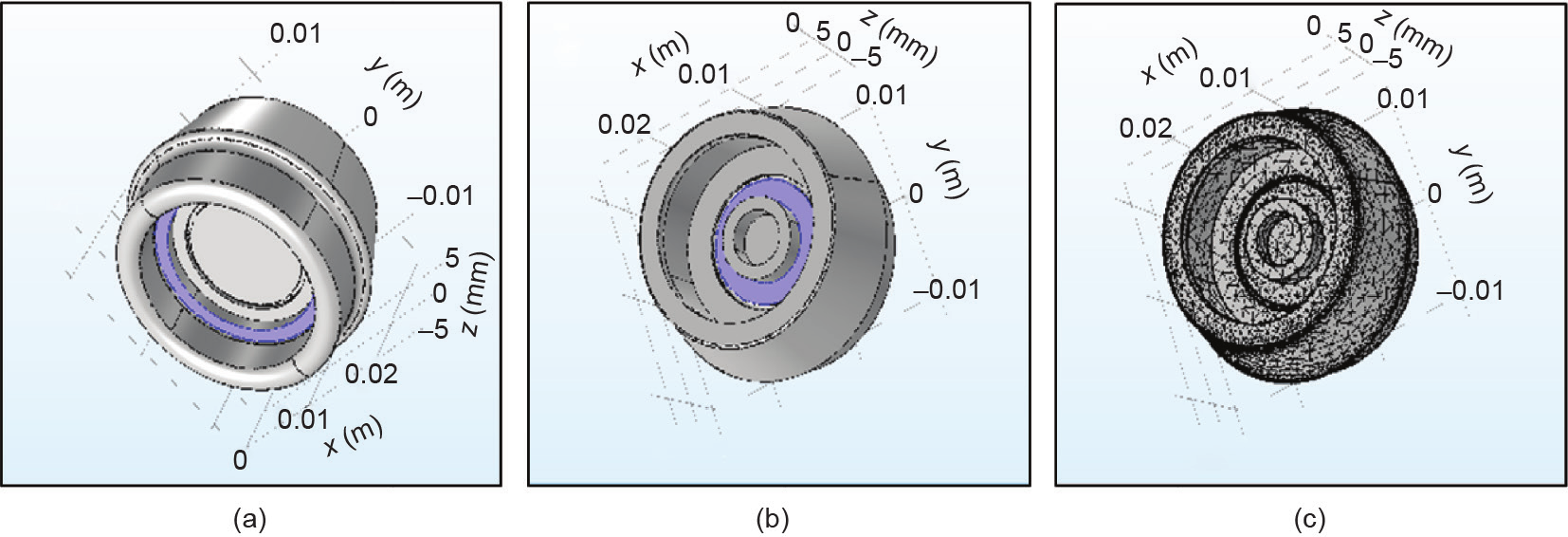
图4. 应力模拟设备的边界约束、载荷和网格元素。(a)边界约束。紫色突出显示的生物传感器外壳边界是用于所有模拟的唯一固定边界约束。其他所有边界都是自由的。(b)边界载荷。紫色突出显示的边界表示传感器激活盖在使用期间施加分布式压力负载的表面。所有模拟都使用100N的总负载,分布在这个突出显示的区域。(c)网格元素。COMSOL预定义的“法向”网格划分用于所有模拟。
《图5》
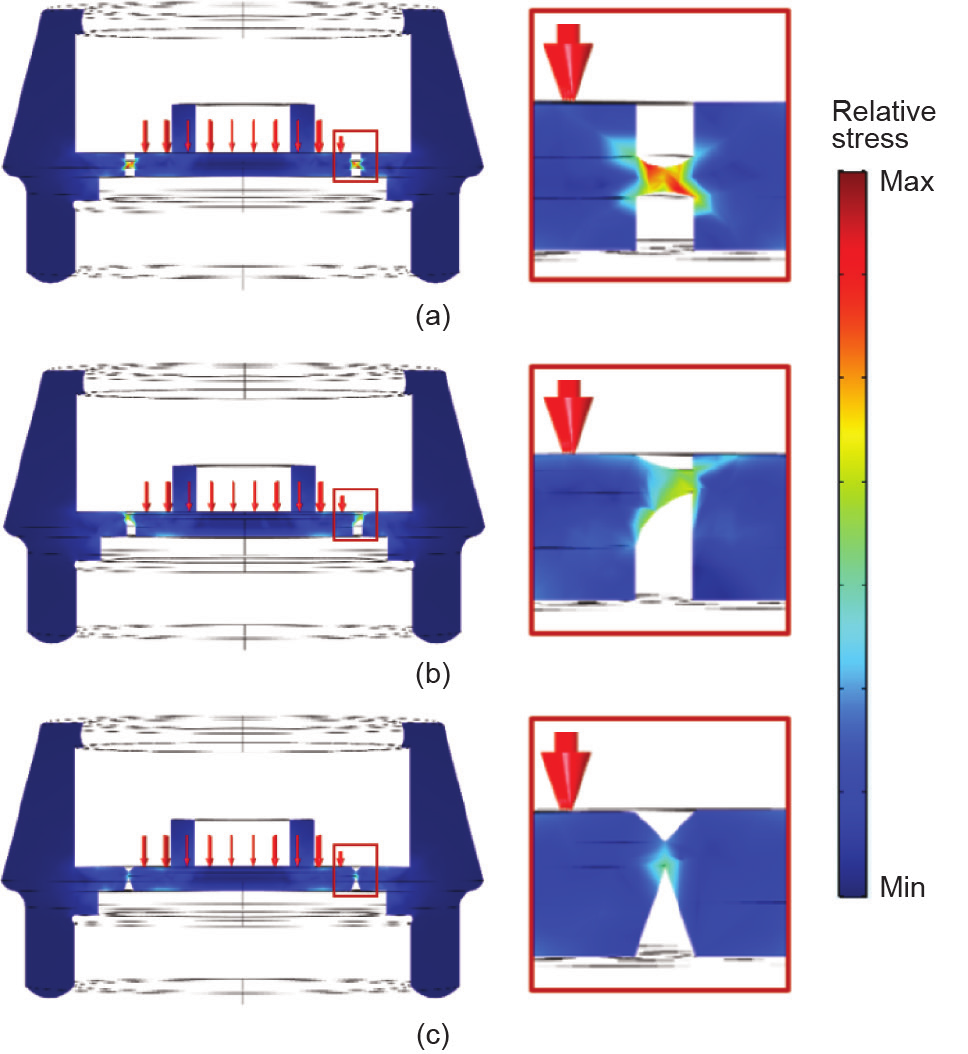
图5. 三种不同设备迭代的横截面与米塞斯应力分布。(a)生物传感器外壳的应力聚焦切口部分的最终设计配置和应力分布;(b)弧形到达某一点的替代设计;(c)具有峰值的替代设计。如图所示,最大的应力和最大的高应力分布决定了(a)的最终设计方案。所有模拟材料都具有以下特性:材料密度为1150 kg·m–3 ,杨氏模量为2 GPa,泊松比为0.35。所有模拟使用分布在边界载荷区域的100 N的总载荷。COMSOL预先定义的“正常”网格划分适用于所有模拟。
采用有限元分析方法对设备在使用过程中受力时产生的应力进行数值计算。为了得到最终结果,本文采用COMSOL软件对不同设计的应力聚焦开孔的应力分布进行了建模。通过盖子将均匀分布的压力载荷施加到外壳的表面,以便在切口中产生应力。为了识别给定施加力下切口的应力大小和集中程度,对三种不同的应力聚焦切口进行了建模。与其他初步设计相比,此处给出的最终设计[图3(d)]既具有在给定施加力下切口内产生的最大应力,又具有通过切口的高应力集中的最佳空间分布。切口的这种设计允许我们的设备由相对高强度的材料制成,同时仍然需要相对较小的力通过断开切口来激活传感器。
该设备的设计也易于组装和使用,如图6所示。将40 μL琼脂塞添加到细菌孔中,并在其表面培养上3O-C12活体生物传感器。将装置盖安装到外壳上并连接到培养管上,该培养管将生物样品与环境隔离。然后通过用户的拇指激活设备。盖子上施加的力会在外壳内的应力聚焦切口中产生应力。然后,细菌孔从外壳上脱落并落入样品中。分析过后,可将盖子推入外壳中以形成压合密封的第二阶段,从而在设备倒置或搅动的情况下,防止试管内的样品在倒置或搅拌时泄漏。该密封还允许用户将生物危险废物转移回生物安全实验室以进行适当处置。
《图6》

图6. 设备使用示范。(a)将琼脂添加到生物测定装置内的细菌孔中;(b)将装置盖与装置底座对齐,并用拇指按压,直到压合密封,使用拇指,可以对盖子施加力,释放压配合并将力引导到应力聚焦切口上;(c)然后细菌孔落入样品中,在那里用生物传感器接种样品;(d)使试管内的液体和细菌与环境保持隔离。
浸入样品后,生物传感器培养8 h。然后,3O-C12激活的生物传感器产生mCherry蛋白。如流式细胞术分析[图7(a)]所示,受诱导的生物传感器在590 nm处发射的荧光出现变化,与未受诱导的生物传感器对照荧光峰相比明显出现分离。在剂量-反应实验[图7(b)]中,0.1 μmol·L−1 的3O-C12足以诱导活体生物传感器在生长8 h后在试管中产生可见的荧光。当用流式细胞分析仪定量时,可以观察到传感器的动态范围为0.01~1 μmol·L−13O-C12。
《图7》
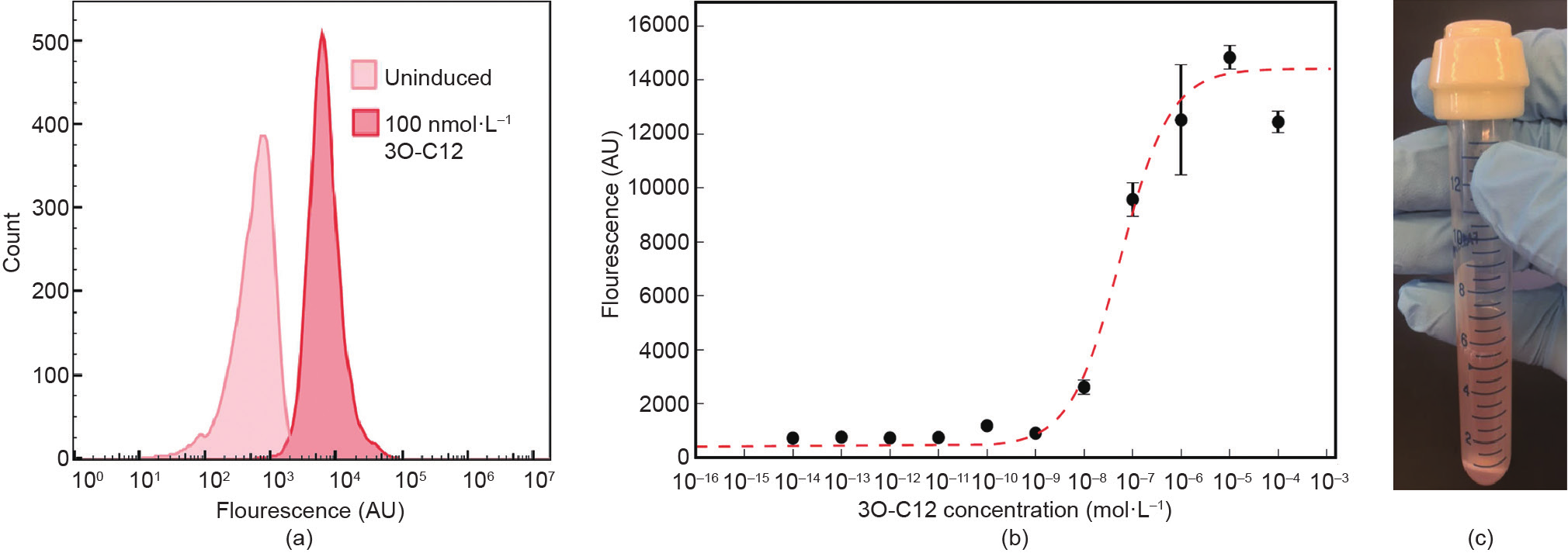
图7. 生物传感器剂量-反应曲线。(a)当用3O-C12诱导时,生物传感器从未诱导状态进入诱导状态。在被诱导后,生物传感器通过mCherry蛋白产生荧光。这些细菌传感器被加入到不同浓度的3O-C12样液中,并维持在指数增殖状态。收集未被诱导的和100 nmol·L−1 3O-C12样品(三个样品)的10 000个事件的数据。(b)诱导8 h后,生物传感器达到mCherry表达的平衡状态,可以量化。然后可以将系统拟合为R2 值为0.987的Hill函数。(c)在足够的时间内,传感器变为拨动开关,并能够输出开/关响应。
《4. 讨论》
4. 讨论
尽管基于合成生物学的生物传感器在检测化学输入方面具有多种用途,但由于受到基因生物的使用和运输法规的限制,许多生物传感器目前仅限于实验室环境。值得注意的是,Pardee等[35]最近开发了一种纸基合成生物传感器。这种基于纸张的生物传感器是通过在纸上冷冻干燥无细胞合成基因网络研制而成的,其目的是产生具有细胞基本转录和翻译特性,但仍然保持非生物性的材料。纸基生物传感器比基于细胞的生物传感器检测速度更快,并且避免了使用活细菌细胞进行检测的缺陷,这使其适合在实验室之外安全地部署工程化基因回路。然而,纸基生物传感器需要不同的样品处理和一组不同的专用材料。因此,我们这里描述的系统允许补充使用活体生物传感器,这可能与未来的无细胞检测方法并行集成。在我们的设想中,我们的系统对缺乏重要医疗基础设施或无法获得医疗保健的地方和人群尤为有益。
3O-C12生 物 传 感 器 能 够 检 测 浓 度 范 围 为0.1~10 μmol·L−1 的3O-C12浓度。相关文献报道表明[36–39],呼吸道中的铜绿假单胞菌感染会引起组织炎症。尽管化学物质3O-C12的具体作用机制尚不清楚,但据信它是通过与细胞表面上的toll样受体相互作用[40],产生炎症反应而产生的。这种炎症是由10~100 μmol·L−1 的3O-C12引发的。受感染肺部的浓度范围在3O-C12生物传感器的动态响应范围之外。然而,可以在分析之前在培养基中直接进行一系列稀释(例如,1/2,1/4,…),如同其他定量测定(如Bradford测定[41])一样。稀释培养基中收集的痰液还可以使细胞更快地分裂,并使其具有更适合生长的环境。
生物测定设备可以在任何3D打印机可用的地方进行制造。我们用于本研究的3D打印机售价不到2000美元,许多新的3D打印机的价格从200美元到500美元不等。我们的生物测定设备也可以根据需要快速制造,并且在2 h内准备就绪[图8(a)]。此处描述的3D打印设备的材料成本约为0.50美元,使用8 g ABS制造。与生物传感器外壳和盖子相比,生物传感器本身仅占设备总成本的一小部分。打印机可以在任何地方使用,这使该设备的部署更加灵活。生物传感器也可以很容易地在世界范围内被培养、冷冻干燥和运输。将少量冷冻干燥的生物传感器装入设备[图8(b)]后,即可密封并准备使用。然后可以将这些装置直接运送到现场以供使用[图8(c)]。在发生设备与环境接触的事故的情况下,重要的是量化设备将生物传感器与环境隔离的程度。使用营养丰富的培养基[图8(d)]来模拟最坏情况。当在100 mL培养基[图8(e)]中以140 r·min–1 摇动时,已经引入培养基的细菌孔能够立即繁殖细胞。当整个完整装置浸没在培养基中时,密封设备能够将生物传感器与环境隔离14 h,然后细胞逸出,并在100 mL培养基中显示出可见的浊度。
《图8》

图8. 从打印机到患者,再到实验室。(a)使用3D打印机打印生物测定设备。(b)在3D打印生物测定设备中培养基因工程传感器。(c)将带有传感器的设备运输至患者所在位置。(d)该装置是防水的,因此,如果该装置落入液体中,则有时间在无GMO逃逸的情况下将其移除。(e)当装置正常使用时,细菌直接沉积在营养丰富的培养基中,它们会立即开始生长。然而,在装置操作不当导致环境液体浸没的情况下,密封装置也可以防止液体接触细胞14 h。为密封和未密封的三个样品收集了10 000个事件的数据。
现场可采集患者痰液样本[图9(a)],并用细胞培养基稀释到样品管中。为了确保样品中没有任何细菌超过卡那霉素耐受的活体生物传感器,可以添加卡那霉素抗生素,使患者痰液中的细菌样本无法存活。然后,将生物传感器的盖子牢固地按压并启动,迫使冷冻干燥的细菌沉淀物进入痰液样品。接下来,可以在现场进行培养和分析[图9(b)]。当维持指数状态时,生物传感器可以同时量化3O-C12暴露的特定水平,或者只需将生物传感器添加到样品中就可以切换到开/关行为[图9(c)]。9 h的孵育可以使生物传感器将液体样品明显地变成红色,从而作为一个双状态系统,有助于在现场直观地确定患者是否感染了铜绿假单胞菌。
《图9》

图9. 与3O-C12接触时的生物传感器活性。(a)从患者身上取出痰样,并将3D打印装置折叠,将细菌释放到样品中。(b)生物传感器的构建可实现3O-C12浓度的量化和更简单的开/关行为,其中3O-C12在10 nmol·L–1 及以上时将使样品明显变红。(c)与初始荧光值F/F0 相比,暴露于诱导物10 h后,生物传感器明显变红,荧光增加98倍。只要细胞仍然暴露于诱导物中,mCherry蛋白就会持续产生。针对未被诱导的和100 nmol·L–1 3O-C12样品(三个样品)收集10 000个事件的数据。
该设备旨在让人员或志愿者在最少的培训下制造和使用。在连接到培养管之后,可以用手抓住设备和培养管,并且拇指可以用于横向夹持以激活传感器。成人使用常用手所产生的平均横向挤压强度为5.0~7.5 kgf(注:1 kgf=9.806 65 N)[42],足以使用该设备。由于此处提供的应力聚焦切口的设计,成人产生的侧向夹紧力将使设备在现场可靠地工作。
《5. 结论》
5. 结论
本文的实验结果表明,当存在铜绿假单胞菌生物膜时,3O-C12生物传感器能够检测诱导物浓度,如在重复感染的情况下。可用于检验目的的活体生物传感器由于封闭在生物测定设备内,因此有可能面临更少的监管障碍,这就允许其在实验室外部署。3D打印设备将GMO转变为整个设备的组件,并确保生物传感器与外部环境隔离。因此,3D打印设备使我们的生物传感器具有处理环境输入的多功能性,使其可作为一种新颖、经济高效的诊断技术进行现场部署。
《Acknowledgements》
Acknowledgements
The authors acknowledge support from funding from federal agencies of the United States including, the National Science Foundation (1709238), as well as funding from Office of Naval Research (N00014-17-12306 and N00014-15-1-2502), and the Air Force Office of Scientific Research (FA9550-13-1-0108). No funding agencies played a significant role in the study design.
《Author contributions》
Author contributions
Wolozny and Ruder conceived the idea for the system described here. All authors designed and performed the experiments, analyzed the data, discussed the results, wrote this manuscript and commented on the paper. Wolozny, Lake, Long, and Ruder revised the manuscript.
《Compliance with ethics guidelines》
Compliance with ethics guidelines
Daniel Wolozny, John R. Lake, Paul G. Movizzo, Zhicheng Long, and Warren C. Ruder declare that they have no conflict of interest or financial conflicts to disclose.














 京公网安备 11010502051620号
京公网安备 11010502051620号




