《1. 引言》
1. 引言
细菌耐药性问题是21世纪人类和动物面临的最严重健康威胁之一,多重耐药(multidrug-resistant, MDR)细菌很有可能导致人类回到无抗生素时代[1‒2]。因此,高度耐药的革兰氏阳性菌(如葡萄球菌、链球菌和肠球菌等)引起的感染被认为是目前重大的公共卫生问题[3],特别是耐甲氧西林的金黄色葡萄球菌(methicillin-resistant Staphylococcus aureus, MRSA)、耐万古霉素的屎肠球菌(vancomycin-resistant Enterococcus faecium, VRE)和耐β-内酰胺的肺炎链球菌的出现,给临床治疗造成了极大的困难。
目前,在临床应用中有超过一半的抗生素,作用靶点是细菌的核糖体,这些抗生素可以通过抑制核糖体30S亚基或50S亚基来阻止细菌蛋白的合成[4‒6]。目前,一些靶向核糖体的抗生素[如大环内酯类、截短侧耳素类(pleuromutilin)、林可酰胺类、链霉素类和恶唑烷酮类]可以作为人用抗生素的替代药物和抗感染治疗的最后防线药物(附录A中的表S1)[2,7‒8]。截短侧耳素类抗生素是一种双萜烯类抗生素,自1951年首次由澳大利亚科学家发现以来,其两种改造衍生物泰妙菌素和沃尼妙林被广泛应用于畜禽养殖领域;2007年和2019年,FDA又批准了可用于人医临床的两种新型截短侧耳素类抗生素:瑞他妙林(RET)和来法莫林(LEF),用于治疗革兰氏阳性菌引起的皮肤和肺部感染[9‒10]。截短侧耳素类抗生素主要作用于细菌核糖体50S大亚基的23S rRNA上,抑制氨基酰tRNA的进位和肽酰转移酶的催化作用,影响肽链合成和延长[10‒14]。
越来越多的证据表明,革兰氏阳性病原体对截短侧耳素的耐药性正逐渐加重,值得关注的是,大多数分离株对截短侧耳素类、林可霉素类(lincosamide)及链阳菌素A类(streptogramin A)具有交叉耐药性,称为PLSA型耐药[15‒18]。对截短侧耳素类的耐药性通常归因于23S rRNA的内源性突变或耐药基因的水平转移,如rRNA甲基转移酶Cfr或抗生素抗性ATP结合蛋白ABC-F家族Vga/Lsa/Sal/VmlR [4,7,19]。ABC-F家族耐药基因存在于多种革兰氏阴性菌和革兰氏阳性菌中,可以引起细菌对靶向核糖体抗生素的广泛性耐药[20‒22]。不同于其他ABC家族耐药基因的外排泵机制,ABC-F家族蛋白并没有跨膜结构,因此关于ABC-F家族耐药蛋白的作用机制,曾经存在很大争议。通过对MsrE、VmlR晶体结构的分析,ABC-F家族耐药蛋白可以通过核糖体保护机制,与抗生素竞争性结合核糖体,导致细菌耐药[23‒25]。
猪链球菌(Streptococcus suis)是一种重要的人畜共患病原体,2005年,中国暴发了规模最大的人类感染猪链球菌的疫情(共造成204例感染、38例死亡)[26]。研究表明,猪链球菌可能作为耐药基因的储存库,促进耐药基因向其他链球菌的传播[27‒28]。在中国,对猪链球菌耐药性的监测研究中,发现了42株泰妙菌素的耐药菌株不含有已知的耐药基因。本研究中,我们鉴定了一种新的耐药基因srpA(猪链球菌核糖体保护性ABC-F家族蛋白),并分析了其理化性质和耐药分子机制。本研究通过功能性克隆、同源建模、分子对接、点突变、药物胞内蓄积试验和核糖体结合分析等对SrpA耐药机制进行了详细阐述。
《2. 材料与方法》
2. 材料与方法
《2.1. 菌株来源》
2.1. 菌株来源
2018年,从中国5个省份共采集166株猪链球菌,其中,72株对泰妙菌素具有耐药性。大肠杆菌BL21、DH5α、金黄色葡萄球菌RN4220以及猪链球菌SD1BY15作为克隆宿主。
《2.2. 生物信息学与序列分析》
2.2. 生物信息学与序列分析
所有样品菌株的基因组DNA均按文献描述的方法进行提取[29],并进行全基因组测序。通过泛基因组分析流程Roary对42个耐药菌株(不含已知耐药基因)和94个敏感菌株进行了全基因组分析[30]。使用Python数据包Pandas对数据进行处理,并提取了10多个分离株中被注释为假定ABC转运蛋白ATP结合蛋白的开放阅读框。使用Matplotlib和Seaborn将数据结果可视化。通过neighbor-joining方法在MEGA7.0中生成ABC-F家族蛋白的系统发育树(bootstrap:1000次)[31]。使用在线工具生成相应蛋白质的序列标识[32]。srpA和上下游区域的核苷酸序列已保存在GenBank数据库中,登录号为MT550884。
《2.3. 的功能性克隆》
2.3. 的功能性克隆
从HNBY78(登录号为PRJNA61672)的基因组DNA中克隆srpA基因,包括其上游区257 bp和下游区89 bp,然后将其连接到穿梭载体pAM401。通过电转的方式将重组质粒转化到受体菌中。
《2.4. SrpA的同源建模和分子对接》
2.4. SrpA的同源建模和分子对接
使用SWISS-MODEL网络服务器(http://www.swissmodel.expasy.org)进行同源建模,构建了SrpA的三维(3D)模型。srpA与嗜热菌核糖体-MsrE复合体(PDB ID: 5Zlu.1.u)的晶体结构具有最高的序列同源性(30%)。根据GMQE(全局模型质量估计)和QMEAN(定性模型能量分析)得分,模型1被选为最佳模型[33]。从PDB数据库(5TCU)下载金黄色葡萄球菌核糖体结构,从ChemSpider (http://www.ChemSpider.com)中提取沃尼妙林的分子结构。用Smina作分子对接工具探索SrpA、沃尼妙林和核糖体之间的结合。通过聚类分析,共提取了2000个对接构象。对于RMSD临界值,较小的聚类半径10.0 Å和较小的界面临界值10.0 Å可以产生更好的聚类效果,从而获得了最大聚类中的前10个构象,并使用PyMol对其结果进行分析。
《2.5. loop区和ATP水解谷氨酸的突变分析》
2.5. loop区和ATP水解谷氨酸的突变分析
使用附录A的表S2中列出的引物,通过定点突变的方式构建SrpA突变体。所有引物均在SrpA的N端引入了一个6×His标签。对每个突变体的srpA基因全长进行测序,以确保没有发生无关的突变。1:2000稀释抗His-HRP抗体,通过Western blot方法鉴定SrpA不同突变体的表达情况。
《2.6. 药物胞内蓄积试验》
2.6. 药物胞内蓄积试验
按照Wang等[34]所描述的方法进行药物胞内蓄积试验。将金黄色葡萄球菌RN4220、RN4220-pAM401和RN4220-pAM401-srpA过夜培养,并将每种菌液650 μL转移到1.5 mL的离心管中。在37 °C下振荡平衡5 min后,加入终浓度为2 μg∙mL-1或16 μg∙mL-1的沃尼妙林。在37 °C摇床培养30 min,然后放置在液氮中3 min,最后放置在65 °C水浴3 min进行裂解。裂解物以20 000 × g离心5 min;在液相色谱-串联质谱(LC-MS/MS)分析之前,将上清液用水-甲醇(2:1, V/V)稀释100倍,涡旋混匀。LC-MS/MS系统的条件如下:毛细管电压,2.5 kV;离子源温度,150 °C;去溶剂温度,500 °C;锥孔气(N2)流速,50 L∙h-1;去溶剂气(N2)流速,800 L∙h-1。沃尼妙林在正电喷雾电离(ESI)模式下进行分析。优化的MS/MS转变以及特定锥孔电压和碰撞能量如下:沃尼妙林,锥孔电压22 V,m/z 263.0 > 163.9(碰撞能量,22 eV)。自动停留时间用于确保每个色谱峰获得大约15个数据点。
《2.7. 核糖体结合试验》
2.7. 核糖体结合试验
SrpA蛋白及其突变体的表达和纯化方法如Li等[29]所述。简单来说,将srpA连接到pET28a并转化到大肠杆菌BL21(DE3)细胞。将含有srpA的BL21大肠杆菌过夜培养,然后用IPTG进行大规模诱导[23]。采用SDS-PAGE鉴定srpA的表达情况。然后参照说明书上的方法(德国QIAGEN)在Ni2+-NTA亲和树脂上纯化蛋白质[25]。按照Wu等[35]所述的方法,通过差速离心提取金黄色葡萄球菌RN4220中的核糖体。将细胞重新悬浮在4 mL缓冲液A中(10 mmol∙L-1含4 mmol∙L-1 MgCl2的Tris-HCl、100 mmol∙L-1 KCl和10 mmol∙L-1 pH为7.2的NH4Cl)。30 000 × g,4 °C离心15 min弃去细胞碎片,在4 °C条件下,将上清液以100 000 × g离心120 min,形成核糖体颗粒[25]。
沃尼妙林(VAL)和恩诺沙星(ENR)的荧光结合物(示踪剂)按Zhang等[36]所述的方法进行制备。SrpA和核糖体的浓度针对荧光偏振免疫分析(fluorescence polarization immunoassay, FPIA)进行了优化,结果如附录A中的图S1所示。选择1000 nmol∙L-1的核糖体和5 μmol∙L-1的SrpA进行进一步分析。取70 μL荧光偏振反应液A [10 mmol∙L-1 Tris (pH 7.5)、60 mmol∙L-1 KCl、10 mmol∙L-1 NH4Cl、300 mmol∙L-1 NaCl、6 mmol∙L-1 MgCl2、0.1 mmol∙L-1 ATP]、70 μL纯化核糖体(1000 nmol∙L-1)和70 μL VAL-DTAF或ENR-AF进行抗生素-核糖体结合试验,在37 °C下反应30 min。然后将反应混合液在微孔板读取器中摇动10 s,并在室温下测量络合物的荧光偏振(FP)值,λex = 485 nm,λem = 530 nm,cutoff = 515 nm,G因子 = 1,空白对照仅含有缓冲液A,其他测定条件相同[37]。SrpA抑制VAL-示踪剂与核糖体结合的能力评估如下,在加入VAL-示踪剂之前,将核糖体在70 μL的反应混合液中与5 μmol∙L-1 SrpA、SrpA突变体或牛血清白蛋白(BSA)在37 °C下共同预孵育30 min。在竞争性试验中,用VAL-示踪剂预处理核糖体30 min,然后与SrpA共孵育。
《2.8. 基因位置的确定》
2.8. 基因位置的确定
采用S1核酸酶脉冲场凝胶电泳(S1-PFGE)和Southern blot鉴定srpA在猪链球菌中的位置。如Barton等[38]所述方法制备srpA阳性菌株的凝胶块并进行电泳迁移20 h。
将凝胶倒置并放置在凝胶模具上,并将尼龙膜、三张干燥滤纸和6 in(1 in = 2.54 cm)的纸巾放置在凝胶上方16 h。通过PCR反应从猪链球菌HNBY78的基因组DNA中扩增获得srpA探针,然后使用DIG high prime DNA标记和检测试剂盒Ⅰ(瑞士Roche)进行标记。
《3. 结果》
3. 结果
《3.1. 猪链球菌中一种新型截短侧耳素耐药基因的鉴定和表征》
3.1. 猪链球菌中一种新型截短侧耳素耐药基因的鉴定和表征
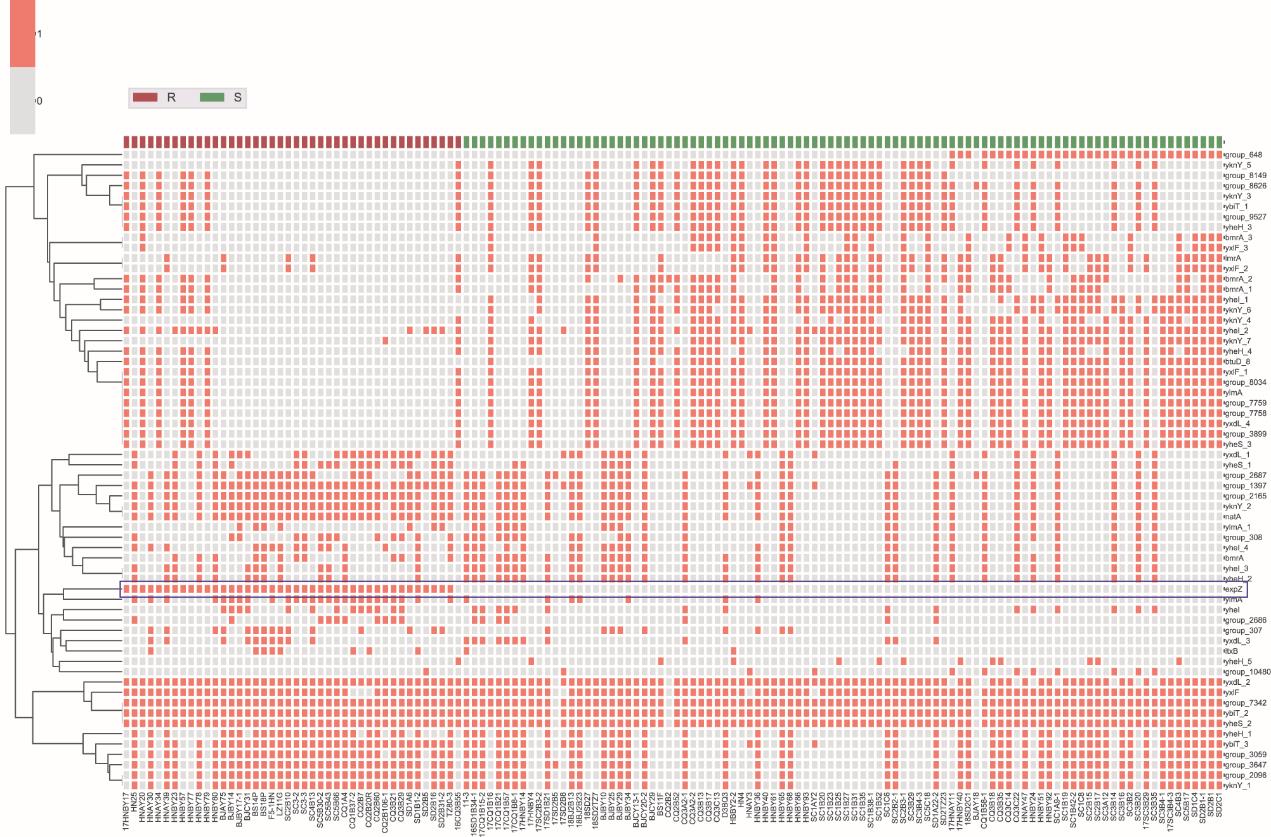
在中国猪链球菌耐药性常规监测期间(2017—2018年),从5个省份共采集166株猪链球菌,其中,泰妙菌素敏感株94株,耐药株72株(64~128 μg∙mL-1)。全基因组测序分析表明,30株耐泰妙菌素菌株中存在PLSA耐药基因lsa(E);然而,在其他42株菌株中未检测到已知的耐药基因。因此,我们选择这42株菌深入分析截短侧耳素耐药的分子机制。利用全基因组分析比较了泰妙菌素敏感株和耐药株之间的基因差异,重点关注了被注释为ATP结合蛋白的开放阅读框(ORF),这些ORF与截短侧耳素耐药性有关[20]。如热图(图1)所示,在所有带有注释的菌株中观察到68种假定的ATP结合蛋白。大多数ORF与截短侧耳素的耐药性无显著相关性。然而,仅在耐药菌株中检测到注释为“expZ”的ORF。然后,我们使用RAST服务器手动检查了“expZ”ORF,并在所有42个耐药菌株中确认了1368 bp的ORF。序列比对表明,该蛋白与另一种抗生素抗性ABC-F蛋白Vga(E)的氨基酸序列同源性约为36%,与VmlR(expZ)的氨基酸序列同源性约为31.6% [39]。为了证实这一假定的耐药基因的作用,将包含该ORF及其上下游区域共1980 bp的DNA片段连接到穿梭载体pAM401中,然后通过电转化的方式将重组质粒转化到猪链球菌SD 1B15和金黄色葡萄球菌RN4220中。与携带空质粒的受体菌相比,携带假定耐药基因质粒的受体菌对沃尼妙林、泰妙菌素、瑞他妙林、来法莫林、维吉霉素M1和林可霉素(LIN)表现出更高的最低抑菌浓度(MIC)(表1),对万古霉素、红霉素、卡那霉素、庆大霉素、四环素、利奈唑胺和氟苯尼考(FFC)的MIC无明显差异。这些结果表明,该1368 bp的ORF是一个新的耐药基因,能够介导对截短侧耳素类、林可酰胺类和链霉素A的交叉耐药性。因此,我们将此ORF命名为SrpA(猪链球菌核糖体保护性ABC-F家族蛋白;登录号为MT550884)。
《图1》
图1 假定ATP结合蛋白在泰妙菌素耐药和敏感菌株中的分布。纵坐标上标注了所有的假定ATP结合蛋白。横坐标显示本试验中包含的136株猪链球菌的名称。红色代表耐泰妙菌素菌株,绿色代表敏感菌株。分别由填充或空白方块来表示ATP结合蛋白的存在与否。“expZ”单独使用矩形边框和星号做突出标记。
《表1》
表1 受试抗菌药物对试验菌株的最低抑菌浓度
| Bacterial isolates | MIC (μg∙mL-1) | ||||||
|---|---|---|---|---|---|---|---|
| VAL | TIA | RET | LEF | VGM | LIN | FFC | |
| 32 | 64 | 64 | 64 | 32 | 128 | 8 | |
| 1 | 4 | 1 | 1 | 1 | 8 | 2 | |
| 1 | 4 | 1 | 1 | 1 | 8 | 2 | |
| 32 | 64 | 64 | 64 | 16 | 128 | 2 | |
| 0.125 | 0.25 | 0.125 | 0.125 | 0.5 | 0.5 | 4 | |
| 0.125 | 0.25 | 0.125 | 0.125 | 0.5 | 0.5 | 4 | |
| 16 | 32 | 32 | 32 | 8 | 16 | 4 | |
| 0.25 | 0.25 | 0.25 | 0.25 | 1 | 0.5 | 4 | |
| 0.25 | 0.25 | 0.25 | 0.25 | 1 | 0.5 | 4 | |
| 2 | 2 | 2 | 1 | 1 | 8 | 4 | |
| 0.5 | 1 | 0.5 | 0.5 | 0.5 | 8 | 4 | |
| 2(4) | 2 | 2 | 2 | 0.5 | 4 | 4 | |
| 0.125 | 0.25 | 0.125 | 0.125 | 0.5 | 0.5 | 4 | |
| 4 | 4 | 2 | 2 | 0.5 | 2 | 4 | |
| 0.125 | 0.25 | 0.125 | 0.125 | 0.5 | 0.5 | 4 | |
《3.2. SrpA与其他抗生素耐药性ABC-F蛋白的相关性研究》
3.2. SrpA与其他抗生素耐药性ABC-F蛋白的相关性研究
SMART (http://smart.embl-heidelberg.de/)序列分析预测SrpA具有ABC-F家族蛋白的典型结构:两个ATP结合位点Walker A(残基36~44和303~311)、两个ATP水解位点Walker B(残基98~102和403~407)、两个ABC家族蛋白特有的特征性序列(残基78~83和383~388)、两段H-loop(残基132~437),进一步预测发现,SrpA并没有跨膜区(附录A中的图S2)。此外,对于SrpA的氨基酸序列分析,其共有特征序列是LSGGE,而不是大多数ABC超家族蛋白中的LSGGQ [40]。
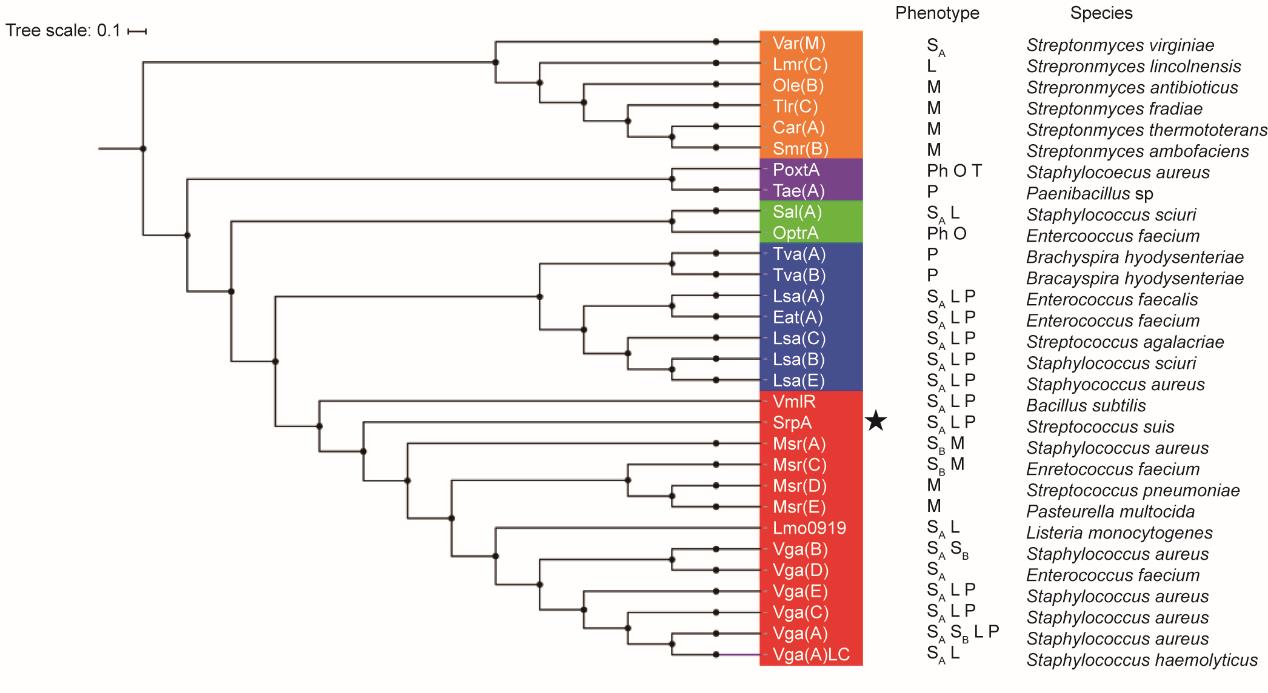
之后,我们采用聚类分析来预测SrpA与其他抗生素耐药ABC-F家族蛋白的关系。如图2所示,所有目前发现的ABC-F家族耐药蛋白可以分为5个大的分支,其中,SrpA主要与Vga家族、Msr家族、发现于单增李斯特菌中的耐药蛋白Lmo0919、发现于枯草杆菌中的VmlR关系较近。序列比对结果显示,SrpA与该谱系的氨基酸同源性超过30%,最接近的同源物是在金黄色葡萄球菌中的Vga(E)(36%)。尽管在多种细菌中都发现了抗生素抗性ABC-F家族蛋白,但SrpA是第一个在猪链球菌中发现的ABC-F蛋白。
《图2》
图2 所有已知的ABC-F家族耐药蛋白的聚类分析。该树代表了1000次重复后获得的一致性结论。从NCBI中获得氨基酸序列。图中标注了不同ABC-F家族蛋白的耐药谱型,此外也标出了在何种细菌中首次发现了不同种的ABC-F家族蛋白。SA:链霉素A;SB:链霉素B;P:截短侧耳素;L:林可酰胺类;M:大环内酯类;Ph:苯酚;O:恶唑烷酮类;T:四环素类;新的抗生素抗性ABC-F耐药因子SrpA用星号表示。
《3.3. SrpA的同源建模和分子对接》
3.3. SrpA的同源建模和分子对接
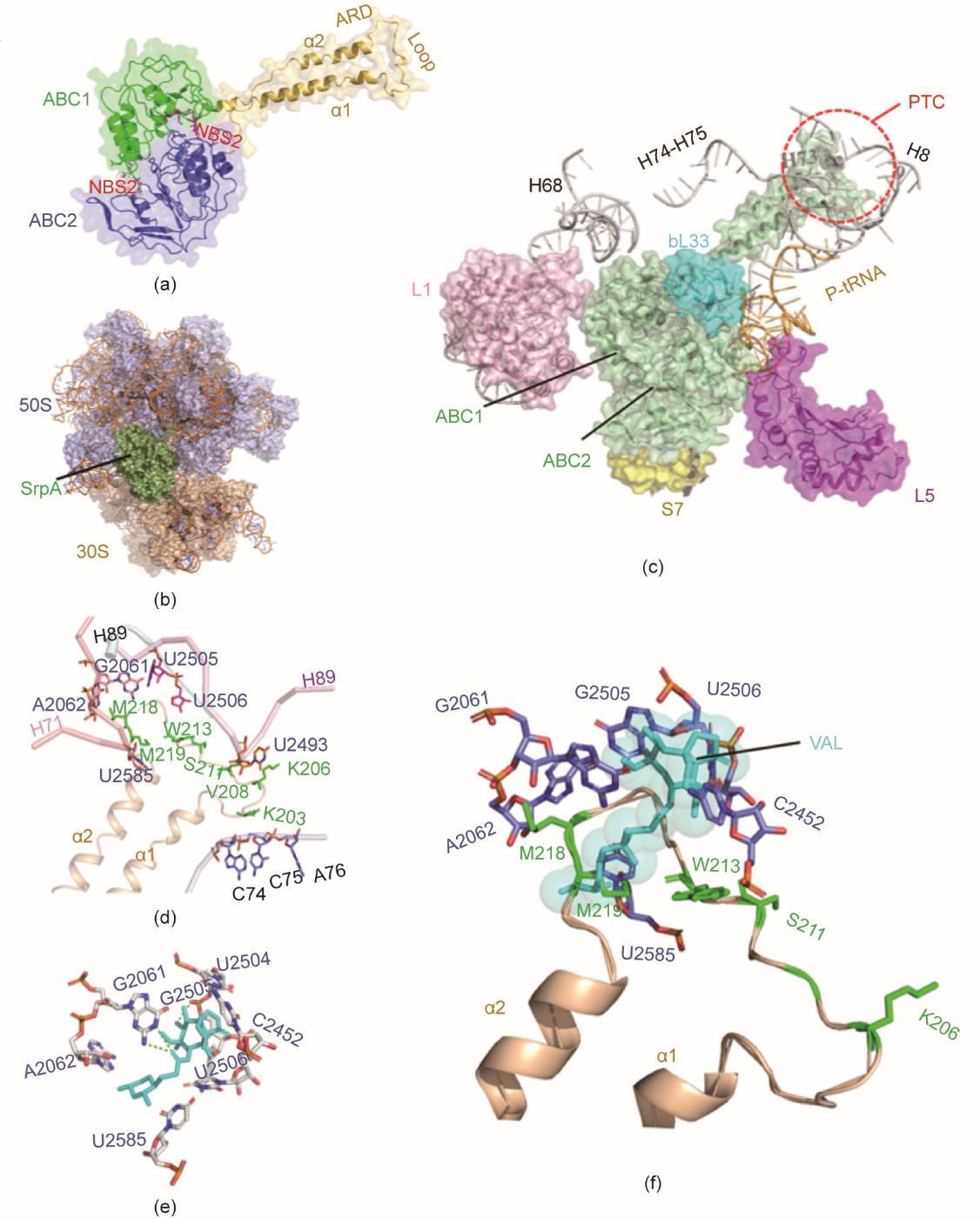
SWISS-MODEL结构预测显示,SrpA与两种已知结构的ABC-F家族蛋白5zlu.1.u和4fin.1.A结构相似度最高(29.85%~25.95%),综合考虑GMQE和QMEAN值,选择根据5zlu.1.u构建SrpA的三维结构。由结构图可知,SrpA具有典型的ABC-F家族蛋白特征,例如,包括两个保守的ABC结构域(ABC1和ABC2)、两个棒状的螺旋linker(163~250),以及一段连接两个linker的loop区(203~224),见图3(a)[20,25]。
《图3》
图3 SrpA的同源建模和分子对接。(a)SWISS-MODEL服务器生成的SrpA的3D结构图;绿色、蓝色和橙色分别标记了二级结构单元ABC1、ABC2和结构域连接蛋白。(b)SrpA(绿色)和核糖体(50S岩灰色,30S浅橙色)的结合模型。(c)SrpA(绿色)ABC1结构域与50S亚基r-蛋白L1(粉红色),bL33(青色),23S rRNA螺旋H68、H73~H75和H89(白色)相互作用;ABC2结构域与30S亚基r-蛋白S7(黄色)、r-蛋白L5(紫色)和tRNA(小麦色)相互作用。(d)SrpA延伸环(橙黄色)和周围关键PTC残基的方向。(e)沃尼妙林核糖体复合体中的沃尼妙林结合口袋(青色)。(f)核糖体-沃尼妙林复合体与SrpA-核糖体复合物的构象重叠。
研究表明,耐药ABC-F蛋白可以与核糖体结合,并以变构方式将药物从其结合位点剥离[23‒24]。在本研究中,采用分子对接分析SrpA和核糖体的相互作用。从PDB数据库(ID: 5TCU)中提取金黄色葡萄球菌70S核糖体的晶体结构,并使用ZDOCK 3.0.2软件,以6°欧拉角采样尺寸对接复合物[图3(b)]。如图3(c)所示,SrpA可以与核糖体结合,SrpA的ABC1结构域面向50S亚基的L1茎,而ABC2结构域与r蛋白L5、S7和P-tRNA的肘部接触。与观察到少数核糖体接触的两个ABC结构域不同,细长的结构域连接蛋白与核糖体建立了广泛的接触,两个linker分别与肽酰-tRNA(P-tRNA)和23S rRNA的H74~H75平行。此外,顶端loop区深入PTC (peptidyl-transferase center)中,与23S rRNA螺旋H89和H73作用。并与P-tRNA的CCA尾端及23S rRNA的C2452、U2505、U2506、A2062、G2061和U2585等多个重要核苷酸接触,而Lys206-Ser224残基及其侧链与U2585、A2062、G2061、U2505、U2506、C2452和U2493核苷酸相互作用[图3(d)]。有趣的是,上述核苷酸似乎构成了一个沃尼妙林的结合口袋,因为沃尼妙林-核糖体对接复合物表明G2061和U2505可以通过氢键固定沃尼妙林的两侧,而其他残基(如U2585、A2062和U2506)也靠近沃尼妙林[图3(e)]。SrpA-核糖体复合物和沃尼妙林-核糖体复合物构象叠加显示,SrpA loop区的219位Met可以直接与结合态的沃尼妙林发生碰撞,产生空间位阻,尤其是Met219残基与沃尼妙林的C14延伸端形成了直接的构象冲突[图3(f)]。
《3.4. 点突变实验验证loop区及ATP水解对SrpA功能的影响》
3.4. 点突变实验验证loop区及ATP水解对SrpA功能的影响
许多研究都证明,loop区的结构和ATP水解能力可以影响ABC-F蛋白的耐药能力,因为ATP水解功能丧失会使EttA (Energy-dependent translational throttle A)不能从核糖体上释放,从而导致ATP水解缺陷的MsrE和Vga(A)显示耐药性降低或消失[23,40‒41]。在本研究中,我们将位于两侧NBD (nucleotide-binding domain)后的保守性催化谷氨酸(NBD1中的Glu103,NBD2中的Glu408)突变为谷氨酰胺。生长曲线显示,这种ATP水解缺陷的SrpA突变体(E103Q/E408Q)对金黄色葡萄球菌的细胞生长没有显著影响(附录A中的图S3)。然而,SrpA (103Q/E408Q)突变体的耐药性显著低于野生型,表明ATP水解对PLSA耐药性至关重要(表1)。
抗生素抗性ABC-F蛋白的loop区统称为抗生素抗性域(antibiotic resistance domain, ARD),因为该蛋白的顶端loop区能够进入核糖体的PTC并直接侵入PTC靶向抗生素的结合位点[21,42]。基于先前的研究和对对接复合体结构的分析可知,SrpA的loop区似乎在发挥耐药作用的效率和特异性中起主要作用。
使用ClustalX对SrpA、Vga同源物、Msr同源物、VmlR和Lmo0919进行多序列比对,发现该短片段对应于SrpA中的残基Lys203~Ser224,为loop区生成了一致的序列标识[图4(a)]。在SrpA中共发现四个高度保守的氨基酸残基:Lys203、Lys206、Gly207和Ala217。氨基酸在其他位点的排列是多态性的,尤其是Val208、Trp213、Met218、Gly220和Ser221。为进一步确认loop区对抗生素耐药性效率和特异性的影响,我们在SrpA loop区的Trp206Ala、Ser211Ala、Trp213Ala、Met218Ala和Met219-Ala位置进行了突变。然而在Lys203、Val208、Gly220和Ser221上进行点突变并不成功,因为这些突变非常不稳定。同样,在Lys203、Val208、Gly220和Ser221这4个氨基酸位点进行的点突变也不成功,因为这几个位点的突变同样会导致突变体稳定性降低。此外,通过同源重组构建了缺失loop区(残基Lys203~Ser224,Δloop)的重组载体pAM401。
《图4》
图4 SrpA介导核糖体保护活性的关键氨基酸。(a)SrpA、Vga(A)、Vga(E)、Msr(E)、VmlR和Lmo0919生成的loop区氨基酸残基序列标识。每个字母的高度与给定位置中相应氨基酸残基的突变频率成正比,x轴为loop区氨基酸的序号。(b)金黄色葡萄球菌RN4220中SrpA和SrpA突变体的Western blot分析。(c)药物胞内蓄积试验(灰色表示用2 μg∙mL-1沃尼妙林处理的样品;蓝色表示用16 μg∙mL-1沃尼妙林处理)。(d)核糖体结合试验。第1列和第2列显示了VAL和ENR-示踪剂的FP值;第3列:向体系中加入纯化的金黄色葡萄球菌核糖体(1000 nmol∙L-1);第4列:核糖体与0.5 μmol∙L-1的SrpA预孵育30 min;在第5~11列中,SrpA被突变体[K206A、S211A、W213A、M218A、M219A、Δ (K203~S224)]或BSA取代;第12列:将1000 nmol∙L-1纯化的金黄色葡萄球菌核糖体添加到ENR-示踪剂系统中;第13列:首先用VAL-示踪剂预处理纯化的金黄色葡萄球菌核糖体30 min,然后将5 μmol∙L-1的SrpA加入反应体系;第14列:5 μmol∙L-1的SrpA直接添加到不含核糖体的VAL-示踪剂中。试验结果为三个独立重复的平均值;误差线代表SD值。没有添加核糖体作为对照。R:金黄色葡萄球菌核糖体。
抗生素药敏试验表明,loop结构域中残基的突变的确影响了细菌的PLSA耐药表型(表1)。与携带完整SrpA的菌株相比,在残基206、211、213、218和219处发生突变的菌株对林可霉素的MIC降低了2~4倍,对维吉霉素的MIC降低了8~16倍。对于截短侧耳素类抗生素,携带Trp206Ala、Trp213Ala或Met219Ala突变的菌株对沃尼妙林、泰妙菌素、瑞他妙林和来法莫林的MIC降低了2~16倍,Ser211Ala位点的突变使MIC降低了16~32倍。有趣的是,loop区(残基Lys203~Ser224,Δloop)的缺失甚至是单个残基(Met218Ala)的突变,可以完全消除其介导PLSA耐药的能力。作为对照,所有突变体对氟苯尼考的耐药性保持不变。综上所述,这些结果表明loop区,尤其是Met218位点,对于细菌PLSA耐药性至关重要。
使用抗6×His的抗体做Western blot分析来检测转化子中SrpA突变体的表达。在含有SrpA及其7种突变体的阳性对照金黄色葡萄球菌RN4220中鉴定到一条分子质量约为55 kDa的单一条带(Trp206Ala、Ser211Ala、Trp213Ala、Met218Ala、Met219Ala、Glu103Gln、Glu408Gln)。携带缺失loop区的srpA转化子显示出大小为53 kDa的蛋白质条带[图4(b)],这种蛋白质分子质量大小的变化可能是由于缺失了loop区所导致的。
《3.5. 药物胞内蓄积试验和核糖体结合试验》
3.5. 药物胞内蓄积试验和核糖体结合试验
本研究通过药物胞内蓄积试验和优化后的核糖体结合试验进一步阐明了SrpA的作用机制。沃尼妙林蓄积的时间过程如图4(c)所示。受试分离株中沃尼妙林的代表性UHPLCMS/MS色谱图展示在附录A中的图S4中。原始受体菌株金黄色葡萄球菌RN4220和携带空pAM401质粒或pAM401/srpA的两个转化子对于沃尼妙林蓄积能力(分别添加2 μg∙mL-1和16 μg∙mL-1的沃尼妙林)表现出相似的趋势,表明SrpA可能不能充当抗生素转运蛋白。
更多的证据表明,抗生素抗性ABC-F蛋白可能是通过核糖体保护机制来介导细菌对抗生素的耐药性,因此我们使用了优化后的FPIA(fluorescence polarization immunoassay)法来检测SrpA和核糖体之间的互作。荧光偏振值(fluorescence polarization, FP)由荧光标记的抗生素示踪剂的分子量决定。未结合的示踪剂,分子量较小,布朗运动较快,荧光偏振值较低,而与大分子结合后的示踪剂,分子量增加,布朗运动较慢,荧光偏振值较高。当体系中仅存在VAL-DTAF或者ENR-AF时,FP值分别为58和66 [图4(d)]。加入1000 nmol∙L-1经过纯化的金黄色葡萄球菌核糖体后,Val-核糖体复合物的FP值上升至160;而ENR复合物的FP值几乎没有变化,表明沃尼妙林和细菌核糖体之间存在特异性结合。
有趣的是,当金黄色葡萄球菌核糖体与SrpA (5 µmol∙L-1)预孵育30 min后再滴定到VAL-DTAF中时,复合物的FP值降低到79左右,远低于直接相互作用的FP值,表明SrpA可以与核糖体相互作用并抑制了其与VAL-示踪剂的结合。先将核糖体与VAL-示踪剂预处理30 min,再加入SrpA (5 µmol∙L-1)共孵育30 min,FP值结果接近88,表明SrpA可以取代与核糖体预结合的VAL-示踪剂。当用BSA取代SrpA预处理金黄色葡萄球菌核糖体时,FP值相似,证实取代沃尼妙林与核糖体的结合是SrpA的一项重要功能。loop区在介导抗生素耐药方面发挥着重要作用,这一结果与FPIA一致。核糖体与SrpA loop区缺失株的共孵育不会降低核糖体相关的VAL-示踪剂的FP水平。此外,我们证实了loop区残基Trp206、Ser211、Trp213、Met218和Met219的重要性,因为位点的Ala突变会导致SrpA的核糖体保护活性显著丧失。
《3.6. 猪链球菌的特性》
3.6. 猪链球菌的特性
srpA阳性猪链球菌来自地理距离较远的5个省份,表明该基因在中国广泛分布。S1-PFGE和Southern blot显示srpA位于所有阳性菌株的染色体DNA中(附录A中的图S5)。根据基因环境分析将分离株分为两类。Types I型流行率最广,包含了40个菌株,一个2078 bp的青霉素结合蛋白基因pbp2b位于srpA基因的下游,一个804 bp的金属依赖型水解酶基因yycJ位于srpA的上游(图5)。有趣的是,这两个基因在猪链球菌呈现高度保守,即使在没有srpA基因的菌株中也是如此,并且,在Type I型基因环境中未能发现插入序列或转座子。Types Ⅱ型分离株在srpA的上游和下游,各有一个反向的插入序列,是两个高度重复的ISSsu8样元件(93%同一性)。反向PCR表明,包含srpA的插入元件可以重组并形成一个3195 bp的环状中间产物,包括srpA和一个ISSsu8样位点的拷贝。
《图5》
图5 猪链球菌中srpA的基因环境。红色为srpA基因标,黑色为ISSsu8-like插入序列,其余部分则标为蓝色,灰色阴影部分代表序列相似度大于90%的序列。从NCBI下载了猪链球菌05ZYH33 (CP000407.1)、98HAH33 (CP000408.1)、P1/7 (AM946016.1)、1081 (CP017667.1)和0061 (CP017666.1),hp (hypothesis protein):假定蛋白。
《4. 讨论》
4. 讨论
自从1929年发现青霉素以来,在兽医临床诊疗中,抗菌药物对于控制细菌感染是必不可少的[43]。然而,由于细菌耐药性的产生,抗菌药物的有效性严重下降。更令人担忧的是,许多感染是由高度耐药的革兰氏阳性菌引起的,MRSA、VRE和产β-内酰胺酶耐药链球菌的出现严重制约了现有抗生素的选择和使用。
为了减少这些多重耐药菌的产生,核糖体靶向抗生素已被开发为与β-内酰胺类或万古霉素联合使用的替代或辅助药物[3]。核糖体是细胞蛋白质的合成工厂,现阶段临床使用的抗生素有一半以上将细菌核糖体作为靶点[6]。尽管它们的化学结构多种多样,但所有靶向核糖体的抗生素都与核糖体的PTC或新生肽链通道区域相互作用,且这些药物具有重叠的结合位点(A位点、P位点和E位点)[4,7,12]。
猪链球菌是养猪业一种重要的人畜共患病原体,可以通过与受感染的猪或猪肉制品密切接触传播[44‒46]。1998年和2005年,中国发生了两次大规模的人类猪链球菌感染疫情,发病率和死亡率都极高[47]。在中国对猪链球菌耐药性的监测研究中,我们发现了一种新的ABC-F蛋白耐药基因srpA。该基因的ORF编码一个由461个氨基酸构成的蛋白质,并在猪链球菌和金黄色葡萄球菌中介导对截短侧耳素、林可酰胺和链霉素A的交叉耐药性。值得注意的是,所有srpA阳性转化子对新批准的截短侧耳素衍生物来法莫林(2019年,FDA)表现出高水平的耐药性。来法莫林是第一个批准用于人全身性治疗的截短侧耳素类抗生素,被认为是MRSA或多重耐药链球菌的潜在治疗选择,因此,金黄色葡萄球菌和猪链球菌对来法莫林的耐药性情况令人担忧。
SrpA与其他耐药性ABC-F家族蛋白的序列比对显示,SrpA与Vga同源物、Msr同源物、Lmo0919和VmlR具有相同的谱系。SrpA的三级结构与MsrE更相似,包括两个由87个氨基酸连接体连接的ATP结合域。此外,SrpA缺乏在VmlR(残基483~547)和Vga同系物(残基460~520)中发现的C端延伸(CTE),这或许是SrpA(461 aa)与其他蛋白质(521~547 aa)相比长度更短的原因[22]。
ATP酶活性对一些ABC-F蛋白家族成员的功能十分重要。EttA中Walker B基序下游的催化谷氨酸位点的突变会通过抑制蛋白质合成进而影响细胞生长,然而在Vga(A)LC和LsaA中,水解失活的突变体能够抑制核糖体的肽基转移酶活性[22,40]。在MsrE中,两个LSGGE基序的谷氨酸定位在AMP-PNP (adenylyl imidodiphosphate)的γ-磷酸附近,可能参与ATP的水解,而谷氨酸突变体在体外试验中表现为核糖体结合减少,在体内表现为阿奇霉素(AZM)抗性[23]。在本研究中,SrpA Walker B(E103/408Q)突变体没有表现出PLSA抗性,这表明ATP的水解对SrpA的活性至关重要。
采用分子对接进一步探究SrpA与核糖体间的相互作用。基于复合体模型的研究显示,SrpA的ABC1和ABC2结构域与核糖体蛋白及其肘部的P-tRNA(peptidyl-tRNA)存在着相互作用,而ABC1和ABC2之间的linker区,尤其是位于23S rRNA螺旋H89和H73附近的loop区,能够延伸到核糖体PTC深处,并与P-tRNA的CCA尾端相互作用。SrpA与核糖体相互作用的结构和构象与MsrE/VmlR非常相似,表明耐药性ABC-F蛋白的结合位点高度保守[23‒24]。当把沃尼妙林与核糖体对接时,23S rRNA上的C2452、U2505、U2506、A2062、G2061、U2585等位点也可以与其三元环及C14侧链形成氢键和范德华力,构成了沃尼妙林的结合口袋[2,13‒14]。由于SrpA loop区上的残基Met218、Met219和Trp213与沃尼妙林分子周围的关键核苷酸A2062、G2061、U2585和C2452直接接触,SrpA loop区的插入可能导致了沃尼妙林结合口袋的构象变化。此外,SrpA中的Met219与沃尼妙林直接竞争,其功能类似于MsrE中的Leu242。
连接两个串联ABC结构域的连接蛋白是ABC-F家族的一个决定性特征,连接体的loop区在核糖体中的PTC附近形成立体特异性接触[20]。与MsrE和VgaALC突变体不同,loop区的缺失完全消除了对截短侧耳素、林可酰胺和链霉素A的耐药性[22‒23]。此外,Lys206Ala、Ser211Ala、Trp213Ala、Met218Ala和Met219Ala突变体在不同程度上也降低了PLSA耐药性。这种差异可能是因为突变减少了SrpA侧链延伸进入PTC的大小,从而消除了与沃尼妙林的空间冲突,并且降低了与SrpA结合后PTC中相对较小的变构构象变化的幅度[20]。
ABC-F蛋白介导抗生素耐药性的机制一直存在争议。部分研究者认为,其可以通过外排泵将药物泵出,部分实验也确实证实了ABC-F族耐药基因可以导致胞内蓄积的抗生素水平降低,而最近的一项研究表明,ABC-F蛋白可以通过与核糖体相互作用并取代与之结合的抗生素来介导细菌耐药性[20,25]。在本试验中,我们观察到srpA的存在对药物胞内的蓄积没有影响。在后续实验中,我们还发现,添加假定的外排泵抑制剂CCCP (carbonyl cyanide m-chlorophenylhydrazone, 10 μg∙mL-1)或利血平(20 μg∙mL-1)对srpA分离株的PLSA抗性没有影响,而在含有过表达氟喹诺酮外排泵基因NorA的对照菌株金黄色葡萄球菌G7中,环丙沙星的MIC值从32下降到4/8(附录A中的表S3)[48]。值得注意的是,核糖体结合试验表明,SrpA可以取代与核糖体结合的抗生素从而阻止二者的结合。总的来说,所有的试验结果都表明,新基因srpA不能发挥抗生素外排泵的功能,而是与其他ABC-F家族蛋白一样通过核糖体保护机制来发挥作用。
NCBI数据库BLASTp搜索显示,SrpA仅发现在猪链球菌中,在中国、美国、英国、越南和荷兰的分离株中分布着大量SrpA样的蛋白质(82%~99%的氨基酸同源性),表明SrpA已在全球范围内传播。令人感兴趣的是,在大多数分离株中,如05ZYH33 (CP000407.1)、98HAH33 (CP000408.1)和P1/7 (AM946016.1),srpA可以分为三个相邻的ORF,分别相隔100~200 bp。第一个ORF(51个氨基酸)与SrpA(残基1~16)的氨基酸同源性为88%;第二个ORF(180个氨基酸)表现出89%的同源性(残基34~212);最后一个ORF(249个氨基酸)显示了81%的同源性(残基218~455)。我们推测,loop区的缺失可以解释这些菌株对截短侧耳素的敏感性。
在本研究中,所有srpA基因都完整地位于染色体上。就基因环境而言,大多数分离株属于Types Ⅰ型(n = 40),其中,srpA总是位于pbp2b (WP_105203009.1)和yycJ (WP_024376691)之间。然而,没有srpA基因的菌株也表现出类似的排列,它们之间的距离为400~500 bp。只有一些介导PLSA抗性表型的ABC-F蛋白被发现是由染色体基因编码的,包括枯草芽孢杆菌中的vmlR、粪肠球菌中的lsa(A)、猪痢疾短螺旋体中的tva(A),以及松鼠葡萄球菌中的lsa(A)和salA [38,49‒51]。Types Ⅱ型分离株的基因环境与Ⅰ型分离株相比有很大差异,在srpA的上下游区域有两个重复的IS481家族ISSsu8样元件,该片段可以与srpA共同形成一个环形中间体。由于猪链球菌被认为是主要链球菌病原体耐药基因传播的储存库,猪链球菌srpA基因种间传播的潜在风险令人担忧[27,52]。在NCBI数据库中,与Types Ⅱ型片段最接近的是来自中国的猪链球菌1081和0061中的区域(CP017667.1和CP017666.1;71%氨基酸同源性),但两者都没有ISSsu8样元件。根据这些发现,我们推测srpA可能来源于猪链球菌,并认为其对PLSA具有先天耐药性。可以想象,在进化的时间尺度上,一些猪链球菌分离株稳定地继承了来自共同祖先的耐药基因,而其他菌株则失去了srpA基因。目前,猪链球菌在临床诊疗中暴露于多种PLSA抗生素的选择压力之下,因此srpA在可移动遗传元件上的出现或许是PLSA敏感的猪链球菌一种新的生存方式。
总之,在本研究中,我们发现了一种新的抗生素耐药性ABC-F家族耐药基因srpA;该基因介导猪链球菌和金黄色葡萄球菌对截短侧耳素、林可酰胺和链霉素A的交叉耐药性。SrpA具有ABC-F家族特有的蛋白质构象,且与Vga(E)的相似性最高。分子对接试验表明,SrpA的耐药结构域(loop区)可以深入肽基转移酶的中心并占据沃尼妙林结合口袋。点突变试验表明ARD loop区介导了抗生素耐药性的特异性与高效性。此外,具有ATP水解功能的残基(103E/408E)在SrpA活性中发挥了至关重要的作用。最重要的是,我们发现SrpA可以保护核糖体并促进抗生素与其结合位点的解离,这表明抗生素耐药性ABC-F家族蛋白也有类似的核糖体保护机制。
















 京公网安备 11010502051620号
京公网安备 11010502051620号




