《1、 引言》
1、 引言
肺炎克雷伯菌(Klebsiella pneumoniae)广泛存在于自然界中,可感染植物、动物和人类[1]等多种宿主,是奶牛临床型乳房炎(clinical mastitis, CM)的主要诱因之一[2]。CM是奶牛养殖过程中最为常见的一种主要由细菌感染引起且治疗成本较高的疾病[3‒4]。通常由革兰氏阴性菌引起的CM比革兰氏阳性菌引起的CM更难治愈[5],平均治疗成本前者为每例211.03美元,后者为每例133.73美元[6]。肺炎克雷伯菌是仅次于大肠杆菌的第二常见的引起奶牛CM的革兰氏阴性菌,但在减奶量、弃奶率、治疗成本、致死率和扑杀率方面,它的危害高于大肠杆菌[7‒8]。鉴于肺炎克雷伯菌感染对奶牛养殖的经济影响,对其种群结构、抗生素耐药性和致病机制的研究尤为重要。
基于gyrA、parC和位于染色体上的β-内酰胺酶基因序列的系统发育分析表明,肺炎克雷伯菌分离株可被分为三个不同但密切相关的种群:肺炎克雷伯菌(K. pneumoniae, KpI)、准肺炎克雷伯菌(K. quasipneumoniae, KpII)和变栖肺炎克雷伯菌(K. variicola, KpIII)[9‒12]。三个种群除都与人类肠外感染有关外,美国患CM奶牛的奶样中也有检测到KpI、KpII和KpIII分离株[13‒14]。然而,传统实验室诊断方法无法可靠地区分肺炎克雷伯菌的三个种群[15‒16],因此对于每个种群对奶牛的真实危害的评估具有很大挑战性。
目前评估肺炎克雷伯菌感染对奶牛影响的研究,大多集中在毒力因子和抗生素耐药性上。几种细菌因子被认为可能促使肺炎克雷伯菌对奶牛的感染,包括与Fe3+转运相关的fec基因、lac操纵子以及与金属(铁、锌和钙等)代谢相关的基因[14,17]。然而,在人类肠道定植和(或)侵袭性感染中起作用的已知致病因子似乎与奶牛CM肺炎克雷伯菌的致病性无关。例如,clbA-Q [编码大肠杆菌素(enterobactin)],iucA-D和iutA [编码气杆菌素(aerobactin)],irp、ybt和fyu [编码耶尔森菌素(yersiniabactin)],iroBCDEN [编码沙门菌素(salmochelin)],mceA-J [编码小菌素(microcin)]以及rmpA和rmpA2 (黏液表型调节因子A)在奶牛CM肺炎克雷伯菌中很少检测到[17]。此外,肺炎克雷伯菌对抗生素治疗反应不佳,由该菌引起的乳房炎可造成重大经济损失[18]。
奶牛源肺炎克雷伯菌分离株的抗生素耐药率在不同地区之间差异很大。在欧洲和美国,患CM奶牛中分离的克雷伯菌属细菌对四环素(5.6%~19.5%)和β-内酰胺类抗生素(0~6.9%)的耐药率较低[19‒20]。中国的一项研究显示,克雷伯菌属细菌对头孢喹啉、卡那霉素、头孢噻呋、多黏菌素B和四环素的耐药率较高(10%~32%)[21]。在欧洲和美国的奶牛中检测到携带多类抗生素耐药基因的肺炎克雷伯菌,包括对β-内酰胺类(blaCTX-M、blaSHV和blaTEM)、四环素类[tet(B)]和喹诺酮类(oqxAB)抗生素产生耐药性的菌株[14,17,22‒23]。然而,中国很少有关于奶牛CM肺炎克雷伯菌耐药基因检测的报道。
《中国乳业统计年鉴2017》的数据显示[24],截至2016年年底,中国约拥有1500万头奶牛,而平均每头奶牛每年因CM造成的相关经济损失为29~135美元[25]。尽管肺炎克雷伯菌是CM的重要诱因,其抗生素耐药性问题严重,但对中国地区奶牛CM肺炎克雷伯菌的种群结构和分子特征知之甚少。这种信息的缺乏限制了研究者对肺炎克雷伯菌对奶牛危害的理解,并阻碍了关键控制点的制定。因此,本研究于2018—2019年间在我国北方地区的三个大型奶牛场连续采集患CM奶牛的牛奶样本,检测肺炎克雷伯菌的流行情况及其耐药性;利用全基因组测序(WGS)和生物信息学方法系统性地研究了北方地区奶牛CM肺炎克雷伯菌的种群结构和分子特征;利用来自人类临床样本的100个KpI和36个KpII-B分离株的全基因组序列,与本研究中获得的奶牛源KpI和KpII-B序列进行比较,从基因组水平上评估牛源及人源KpI和KpII-B菌株之间的关系。
《2、 材料和方法》
2、 材料和方法
《2.1 样本采集及细菌分离培养》
2.1 样本采集及细菌分离培养
2018年1月至2019年12月。在山东、河北和黑龙江三个具有代表性且同属一家乳企、具有相似饲养和管理方式的大型商业奶牛场,采集患CM奶牛的奶样。每个奶牛场有3000~5000头荷斯坦-费里西亚泌乳奶牛,奶牛均散养在栏圈中,以混合饲料喂养,在特定挤奶室挤奶。在挤奶之前,兽医首先对奶牛进行了CM筛查,将具有乳房肿胀、发热、发硬、发红和(或)牛奶呈水状及有薄片、血块或脓液等临床症状的奶牛归为CM疑似病例;然后按照文献[25‒26]中的方法采集病例奶样,即收集有明显乳房炎症状如乳房肿胀、疼痛、发红和(或)乳汁分泌恶化奶牛的牛奶,并置于2~8 ℃的条件下保存。用接种环蘸取约10 μL牛奶样品接种于科玛嘉显色琼脂培养基上,在37 ℃的条件下培养18~24 h。挑取疑似肺炎克雷伯菌的菌落(蓝色菌落)进行复苏培养,用基质辅助激光解吸电离飞行时间质谱或16S rRNA扩增测序的方法进行种属鉴定,16S rRNA引物详见文献[27]。
《2.2 抗生素敏感性试验》
2.2 抗生素敏感性试验
根据美国临床实验室标准化协会(CLSI)的指南,采用微量肉汤稀释法对肺炎克雷伯菌进行药敏试验[28],大肠杆菌ATCC® 25922为质量控制菌株(QC)。受试抗菌药物包括头孢曲松、头孢噻呋、氟苯尼考、庆大霉素、阿莫西林/克拉维酸、卡那霉素、环丙沙星、替加环素、甲氧苄啶/磺胺甲𫫇唑、四环素、美罗培南和多黏菌素。根据CLSI的文件VET08 [28]、M100-S28 [29]和欧洲抗菌药物敏感性试验委员会(EUCAST)指南[30]的有关标准判定分离株对药物的敏感性。MIC50和MIC90分别代表抑制50%和90%细菌生长所需的最低药物浓度。
《2.3 全基因组测序》
2.3 全基因组测序
使用中国美基生物科技有限公司的细菌DNA试剂盒提取细菌的基因组,用KAPA HyperPrep Kit(瑞士罗氏公司)构建DNA文库,委托安诺优达基因科技有限公司使用Illumina HiSeq 2500平台进行150碱基对(bp)的双端测序。用SPAdes软件(版本为3.13.0)对测序数据进行组装,最小覆盖率为50% [31]。除此之外,还从美国国家生物技术信息中心(NCBI)的数据库中下载了100株人源KpI型和36株人源KpII-B型肺炎克雷伯菌的全基因组序列,其中KpI型菌株为分离自2016—2019年的中国临床分离株,而KpII-B型菌株为2015—2020年间收集自中国、巴基斯坦、泰国、美国、墨西哥、希腊、荷兰和尼日利亚的分离株,详见附录A中的表S1。
《2.4 分子特征分析》
2.4 分子特征分析
使用Kleborate(版本0.4.0)软件确定肺炎克雷伯菌的种群,其原理是通过将拼接好的基因组序列与NCBI数据库中的克雷伯菌基因组进行比对[17]。以BIGSdb数据库和毒力基因数据库为参考,使用SRST2软件包对分离株进行已知耐药基因和毒力基因的筛查[32]。采用多位点序列分型(MLST)的方法对分离株进行序列分型(ST)[33],并利用BioNumerics7.0和BURST算法生成基于ST的最小生成树[34]。使用BioNumerics计算辛普森多样性指数来评估基因型的多样性。通过在线网站筛选出携带SHV型超广谱β-内酰胺酶基因的分离株。
《2.5 单核苷酸多态性及系统发育分析》
2.5 单核苷酸多态性及系统发育分析
用基因组草图进行核心基因组分析,并以肺炎克雷伯菌NTUH-K2044的基因组作为参考序列计算分离株核心基因组的单核苷酸多态性(SNP)[17]。利用Harvest软件包(版本为1.1.2)[36]中的Parsnp构建基于核心基因组SNP的邻近法(NJ)系统发育树,并使用iTOL进行可视化。
《2.6 基因组注释和泛基因组分析》
2.6 基因组注释和泛基因组分析
用基因组快速注释工具Prokka对组装好的基因组草图进行注释[37]。将生成的GFF3作为输入文件,使用Roary (version 3.11.2)生成泛基因文件(包括一个gene_presence_absence.csv文件)[38]。使用全基因组关联分析(GWAS)软件Scoary计算细菌附属基因组与宿主性状之间的关联[39]。基于GWAS分析和基因功能注释的结果,评估可能影响肺炎克雷伯菌在奶牛中的适应性和毒力的因素。将在5%~95%的肺炎克雷伯菌基因组中发现的基因定义为常见的附属基因,使用R(版本3.5.3)中的prcomp函数对这些常见的附属基因进行主成分分析(PCA)[17]。
《3、 结果》
3、 结果
《3.1 CM牛奶样品中肺炎克雷伯菌的流行情况》
3.1 CM牛奶样品中肺炎克雷伯菌的流行情况
在三个奶牛场的6301份CM牛奶样本中,共分离到183株肺炎克雷伯菌(附录A中的图S1)。两年间肺炎克雷伯菌的年检出率基本一致(p = 0.97),其中2018年的检出率为3.0%(92/3053),2019年的检出率为2.8%(91/3248)。此外,三个省份之间肺炎克雷伯菌的年检出率也没有显著差异,其中河北省牧场2018年的检出率最高(3.5%, 26/751),黑龙江省牧场2018年的检出率最低(2.5%, 21/831)(图1和附录A中的表S2)。
《图1》
图1 2018—2019年我国北方地区CM奶样中肺炎克雷伯菌的年检出率。
《3.2 肺炎克雷伯菌的种群结构和遗传多样性》
3.2 肺炎克雷伯菌的种群结构和遗传多样性
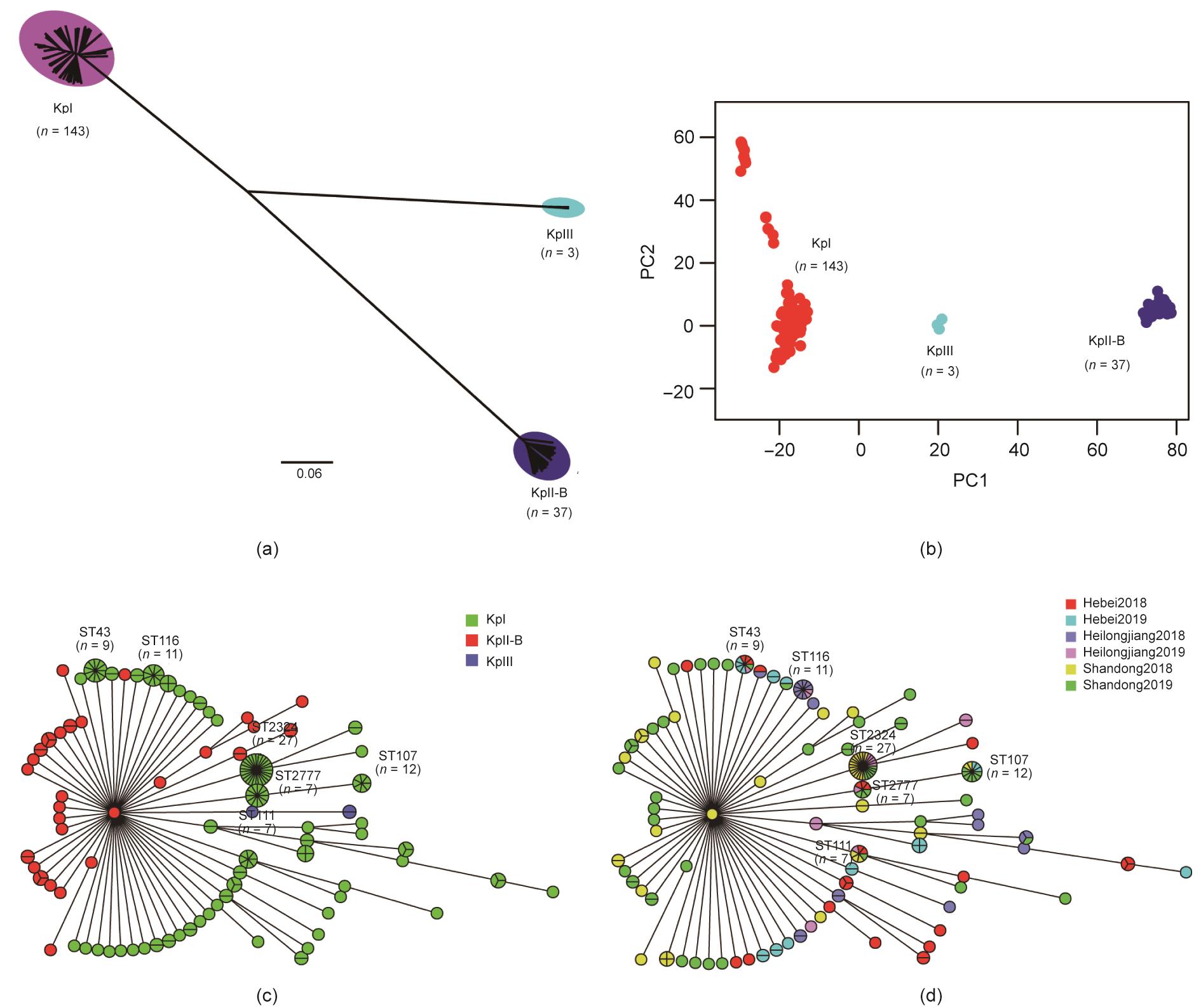
全基因组测序和基于核心基因组SNP的邻接法(neighbor-joining, NJ)系统发育分析表明,183株奶牛肺炎克雷伯菌可分为KpI、KpII-B和KpIII三大不同的种。其中,KpI数量最多(78.1%, 143/183),KpII-B次之(20.0%, 37/183),KpIII最少(1.6%, 3/183)[图2(a)]。种间核心基因的平均核苷酸同源性(ANI)为96.2%~96.6%,而同一种群内的基因组同源性超过99.4%(附录A中的表S3)。对5%~95%基因组中存在的5174个辅助基因进行PCA分析发现,183个基因组依据辅助基因的携带情况,自动聚类成三个群,分别对应KpI、KpII-B和KpIII三个种,进一步证明了三个种的肺炎克雷伯菌都能导致中国奶牛的乳房感染[图2(b)]。
《图2》
图2 奶牛CM源肺炎克雷伯菌分离株的种群结构。(a)基于核心基因组SNP的NJ系统发育树;(b)基于(存在于5%~95%基因组中的)肺炎克雷伯菌辅助基因的主成分分析;(c)KpI型、KpII-B型和KpIII型肺炎克雷伯菌的MLST最小生成树;(d)2018—2019年,各省份肺炎克雷伯菌分离株的MLST最小生成树。
MLST分型结果显示KpI、KpII-B和KpIII具有显著的遗传多样性。143个KpI菌株共检测到50个ST型,辛普森多样性指数为94.2%,37个KpII-B菌株中鉴定出26个ST型,辛普森多样性指数为97.90%(附录A中的表 S4)。虽然KpIII只有三株,但它也有两种ST型(辛普森多样性指数为66.67%)(附录A中的表 S3)。KpI最常见的基因型(n ≥ 5株)为ST2324(18.9%, 27/143)、ST107(8.4%, 12/143)和ST116(7.7%, 11/143),而KpII-B和KpIII各ST型菌株数量基本都少于三株[图2(c)]。通过对我国北方CM奶牛KpI基因型的分布和动态分析,发现在同一牛群中没有稳定的优势ST型。2018年黑龙江牛群中最流行的ST型是ST116(42.9%, 9/21),而2019年最流行的是ST2324(33.33%, 5/15)。2018年,山东省牛群中包含菌株数量最多的ST型为ST2324(55.2%, 16/29),而2019年为ST107(23.3%, 7/30)。河北省牛群中未观察到显著优势基因型,各ST型菌株数量无明显差异[图2(d)]。
《3.3 抗生素敏感性》
3.3 抗生素敏感性
在183株肺炎克雷伯菌分离株中,甲氧苄啶/磺胺甲𫫇唑的耐药率较高(97.3%, 178/183),而四环素(20.2%, 37/183)和头孢噻呋(14.8%, 27/183)的耐药率中等。相比之下,头孢曲松(5.5%, 10/183)、氟苯尼考(5.5%, 10/183)、庆大霉素(2.7%, 5/183)、阿莫西林/克拉维酸盐(1.1%, 2/183)、卡那霉素(1.1%, 2/183)、环丙沙星(0.5%, 1/183)和替加环素(0.5%, 1/183)的耐药率较低,所有菌株对美罗培南和多黏菌素均敏感。然而,三个种的菌株在体外对抗生素的敏感性不同。KpI对12种抗生素的MIC范围分布较KpII-B和KpIII更广。除头孢曲松、头孢噻呋和四环素外,KpI、KpII-B和KpIII对大多数抗生素的MIC50和MIC90相似(表1)。
《表1》
表1 2018—2019年我国北方地区奶牛CM源KpI、KpII-B和KpIII型肺炎克雷伯菌的耐药特征
| Antibiotics | Phylogroups | MIC (μg∙mL-1) | ATCC® 25922 | Resistance rate (%) a | |||
|---|---|---|---|---|---|---|---|
| MIC50 | MIC90 | Range | For | For each phylogroup | |||
Amoxicillin/ clavulanate | KpI | 2/1 | 8/4 | 1/0.5―32/16 | 2/1 | 1.1% (2/183) | 1.4% (2/143) |
| KpII-B | 2/1 | 4/2 | 2/1―8/4 | 0 (0/37) | |||
| KpIII | 2/1 | 4/2 | 2/1―4/2 | 0 (0/3) | |||
| Ceftiofur | KpI | 1 | 8 | 0.25―8 | 0.25 | 14.7% (27/183) | 18.9% (27/143) |
| KpII-B | 1 | 1 | 1―2 | 0 (0/37) | |||
| KpIII | 1 | 2 | 0.5―2 | 0 (0/3) | |||
| Ceftriaxone | KpI | 0.06 | 2 | 0.01―128 | 0.03 | 5.5% (10/183) | 7.0% (10/143) |
| KpII-B | 0.12 | 0.12 | 0.06―0.12 | 0 (0/37) | |||
| KpIII | 0.12 | 0.12 | 0.06―0.12 | 0 (0/3) | |||
| Meropenem | KpI | 0.03 | 0.03 | 0.01―0.06 | 0.03 | 0 (0/183) | 0 (0/143) |
| KpII-B | 0.03 | 0.03 | 0.03―0.03 | 0 (0/37) | |||
| KpIII | 0.03 | 0.03 | 0.03―0.03 | 0 (0/3) | |||
| Gentamicin | KpI | 0.25 | 0.5 | 0.06―64 | 0.25 | 2.7% (5/183) | 3.5% (5/143) |
| KpII-B | 0.25 | 0.5 | 0.25―1 | 0 (0/37) | |||
| KpIII | 0.25 | 0.5 | 0.25―0.5 | 0 (0/3) | |||
| Kanamycin | KpI | 1 | 1 | 0.25―128 | 1 | 1.1% (2/183) | 1.4% (2/143) |
| KpII-B | 1 | 1 | 1―2 | 0 (0/37) | |||
| KpIII | 1 | 1 | 1―1 | 0 (0/3) | |||
| Tetracycline | KpI | 4 | 64 | 1―64 | 1 | 20.2% (37/183) | 25.9% (37/143) |
| KpII-B | 4 | 4 | 1―8 | 0 (0/37) | |||
| KpIII | 4 | 4 | 4―4 | 0 (0/3) | |||
| Tigecycline | KpI | 0.5 | 1 | 0.25―8 | 0.25 | 0.5% (1/183) | 0.7% (1/143) |
| KpII-B | 0.5 | 1 | 0.25―1 | 0 (0/37) | |||
| KpIII | 0.5 | 0.5 | 0.5―0.5 | 0 (0/3) | |||
| Ciprofloxacin | KpI | 0.03 | 0.06 | 0.01―8 | 0.01 | 0.5% (1/183) | 0.7% (1/143) |
| KpII-B | 0.06 | 0.06 | 0.01―0.12 | 0 (0/37) | |||
| KpIII | 0.03 | 0.06 | 0.03―0.06 | 0 (0/3) | |||
| Florfenicol | KpI | 4 | 4 | 2―128 | 4 | 5.5% (10/183) | 5.6% (8/143) |
| KpII-B | 4 | 4 | 2―16 | 5.4% (2/37) | |||
| KpIII | 4 | 4 | 2―4 | 0 (0/3) | |||
| Colistin | KpI | 1 | 2 | 1―2 | 0.5 | 0 (0/183) | 0 (0/143) |
| KpII-B | 1 | 2 | 1―2 | 0 (0/37) | |||
| KpIII | 1 | 1 | 1―1 | 0 (0/3) | |||
Trimethoprim/ sulfamethoxazole | KpI | 8/152 | 8/152 | 0.25/4.75―8/152 | 0.25/4.75 | 97.2% (178/183) | 96.5% (138/143) |
| KpII-B | 8/152 | 8/152 | 8/152―8/152 | 100% (37/37) | |||
| KpIII | 8/152 | 8/152 | 8/152―8/152 | 100% (3/3) | |||
《3.4 耐药性基因和毒力基因》
3.4 耐药性基因和毒力基因
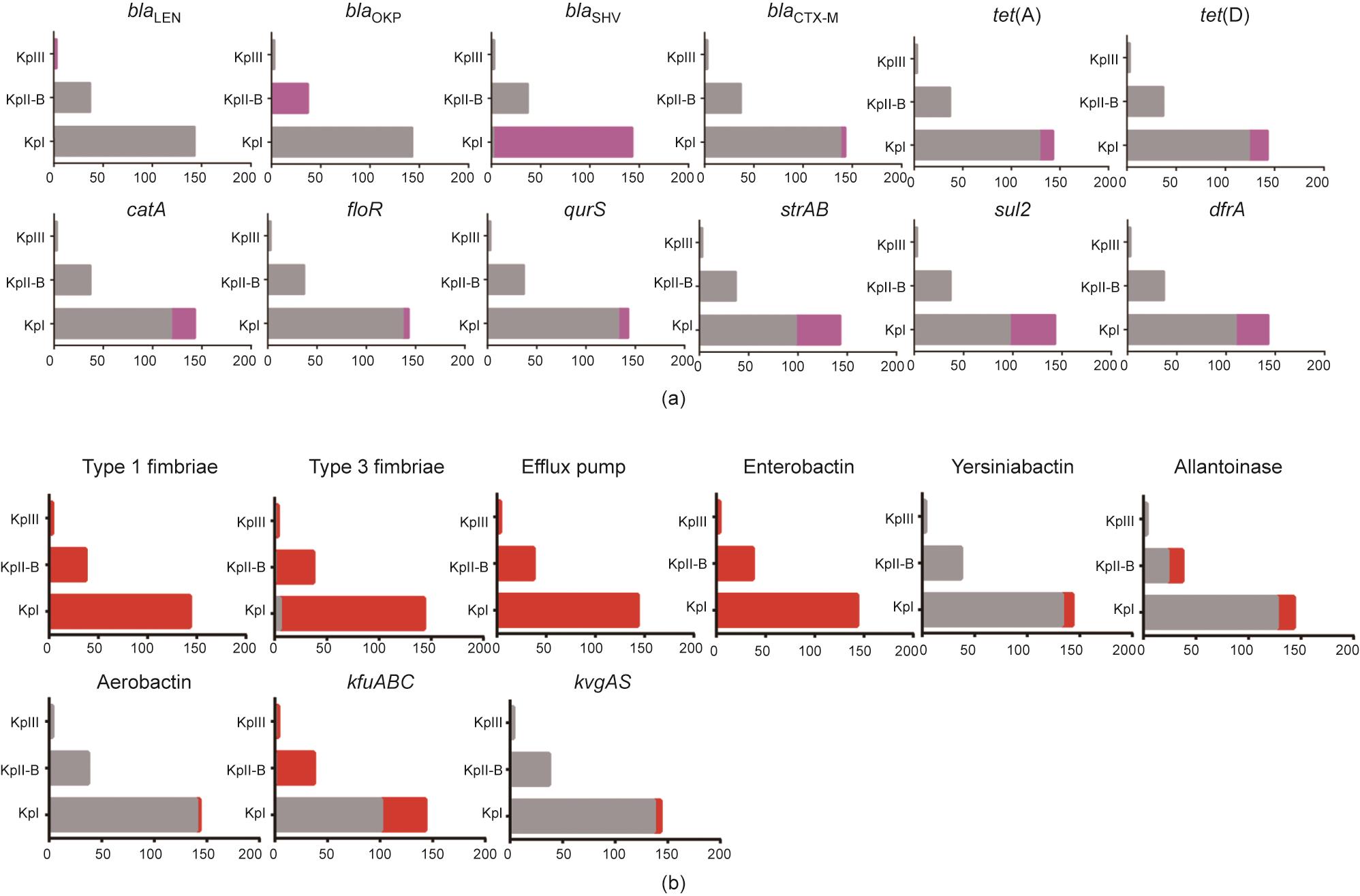
在183个分离株中共检测到57种耐药性基因(附录A中的表S5),其中,78.9%(n = 45)的耐药基因由KpI携带。在KpI型肺炎克雷伯菌中,β-内酰胺类耐药基因类型最丰富,100%的菌株携带blaSHV(21.0%属于产生ESBL型blaSHV变体,79.0%属于不产生ESBL型blaSHV变体),3.5%携带blaCTX-M(blaCTX-M-14和blaCTX-M-15),2.8%携带blaTEM-1。在KpI型肺炎克雷伯菌中氨基糖苷类耐药基因strAB、氟苯尼考耐药基因catA和floR、磺胺类耐药基因sul2、甲氧苄啶耐药基因dfrA以及四环素耐药基因tet(A)和tet(D)的检出率(4.2%~32.2%)相对较高(附录A中的表S5)。相比之下,KpII-B和KpIII分离株分别只携带β-内酰胺类耐药基因blaOKP-B和blaLEN [图3(a)]。此外,还发现27株携带ESBL编码基因blaSHV-2a (n = 22)、blaCTX-M-14 (n = 2)或blaCTX-M-15 (n = 3)的KpI菌株对头孢噻呋表现出高度耐药性。
《图3》
图3 奶牛CM源KpI (n = 143)、KpII-B (n = 37)和KpIII (n = 3)分离株的基因组特征。(a)代表耐药基因;(b)代表毒力因子。柱状图中粉色或红色代表该谱系携带该基因的菌株数量,而灰色代表该谱型不携带该基因的菌株数量。
在183个分离株中共到70种毒力因子(附录A中的表S5)。1型和3型菌毛是主要的黏附结构,AcrAB外排泵被认为是对抗宿主固有免疫防御机制的新型毒力因子,三个种群肺炎克雷伯菌都普遍(100%)携带这些毒力。然而,1型菌毛调节基因fimK在所有KpI和KpIII分离株中均被检测到,但在KpII-B分离株中缺失。铁载体系统是肺炎克雷伯菌的关键毒力因子,它可以帮助细菌从环境中获取有限的铁离子,包括大肠杆菌素、耶尔森菌素、沙门菌素和气杆菌素。大肠杆菌素普遍存在于KpI、KpII-B和KpIII的所有分离株中,而气杆菌素(iucA-D和iutA,2.1%)和耶尔森菌素(irp、ybt和fyu,7.0%)仅在KpI分离株中检测到。二元调控系统基因kvgAS也仅在KpI分离株中发现(4.2%)。在37.8%的KpII-B分离株和11.2%的KpI分离株中发现了尿囊素利用基因(allantoin utilization,allABCDRS、ylbEF、glc、fdrA和ybb等)。所有的KpII-B和KpIII菌株中都检测到铁摄取操纵子kfuABC,而KpI菌株的携带率只有29.4% [图3(b)]。
《3.5 奶牛源与人源KpI肺炎克雷伯菌的分子特征及系统发育分析》
3.5 奶牛源与人源KpI肺炎克雷伯菌的分子特征及系统发育分析
为了在基因组水平上确定牛源与人源KpI肺炎克雷伯菌分离株之间的关系,并评估牛KpI肺炎克雷伯菌对人类健康的风险,本研究基于243个KpI基因组中约3 266 330个核心基因组SNP构建了一个NJ系统发育树,其中包括从公共数据库下载的来自中国地区的100个人源KpI和本研究分离的143个牛源KpI。系统发育分析表明,人源和牛源KpI肺炎克雷伯菌呈现出与基因型(特征)一致的深度分支且复杂多样的种群结构特征[图4(a)]。总体而言,奶牛源KpI比人源KpI菌株表现出更高的遗传多样性,MLST分型的辛普森多样性指数分别为94.2%和40.4%。虽然人源和牛源KpI没有形成可区分的宿主特异性谱系,但它们的种群结构存在明显的差异,其中,(中国北方地区患CM的)牛源KpI菌株的常见克隆型为ST2324、ST107、ST116、ST43、ST111和ST2777,而KpI在(中国地区)人医临床的主要发病克隆为ST11、ST23和ST25。ST11是人类KpI分离株中的优势克隆型(63%, 63/100),但牛分离株中尚未出现一致的优势克隆型。此外,虽然在人类和奶牛中都发现了ST661、ST15和ST37型KpI分离株[图4(a)],但它们之间有2300~15 051个核心基因组SNP。
《图4》
图4 人源(n = 100)和CM奶牛(n = 143)KpI分离株基因组特征的比较分析。(a)耐药基因;(b)毒力因子。柱状图中粉色或红色代表该谱系携带该基因的菌株数量,而灰色代表该谱型不携带该基因的菌株数量。
对243个基因组中的辅助基因进行了比较分析,发现4432个常见辅助基因存在于5%~95%的基因组中。进一步的PCA分析表明,仅基于辅助基因分析,无法准确地将奶牛源与人源KpI菌株区分开。然而,人源ST11分离株与奶牛源ST2324分离株之间的辅助基因存在显著差异[图4(b)]。此外,在ST2324菌株中检测到固氮相关基因(nif操纵子),表明它们可能来自植物[17,40]。然后,根据GWAS分析结果和毒力基因检测结果,确定了奶牛源或人源KpI分离株的特有基因。共有654个基因被确定与牛源KpI相关(OR > 1),1154个基因被发现与人源KpI相关(OR < 1)(附录A中的表S5)。其中,人源KpI肺炎克雷伯菌中编码耶尔森菌素(irp、ybt和fyu)、气杆菌素(iucA-D和iutA)、大肠杆菌素(clbA-Q)、沙门菌素(iroBCDEN)、小菌素(mceA-J)等铁载体相关基因,以及rmpA和rmpA2等荚膜调控基因的分离率均显著高于奶牛乳房炎源KpI型肺炎克雷伯菌(p < 0.01)。而在奶牛乳房炎源KpI肺炎克雷伯菌中clpC、lpfA、kfuABC、lac操纵子基因(lacI、lacZ和lacY)和Fe3+转运蛋白相关基因(fecABDEIR)的检出率显著高于人源KpI肺炎克雷伯菌。这些独特的基因可能有利于宿主入侵和适应,以及逃避宿主免疫反应[图4(c)]。
《3.6 奶牛源与人源KpII-B肺炎克雷伯菌的分子特征及系统发育分析》
3.6 奶牛源与人源KpII-B肺炎克雷伯菌的分子特征及系统发育分析
由于我国人医临床分离的KpII-B肺炎克雷伯菌数量较少(n = 11),对比分析时从NCBI数据库中下载了来自巴基斯坦(n = 8)、泰国(n = 2)、美国(n = 5)、墨西哥(n = 1)、希腊(n = 1)、荷兰(n = 1)和尼日利亚(n = 7)的25个基因组序列一起进行研究。人源和牛源KpII-B肺炎克雷伯菌系统发育分析结果与KpI分离株相似,在核心基因组SNP系统发育树上未出现明显的宿主特异性谱系[附录A中的图S2(a)]。来自人类临床的一株KpII-B(09A323)和来自奶牛的两株KpII-B(SD130-19和SD52-19)表现出高度的核苷酸同一性,它们之间存在326个核心基因组SNP(326/3296574,占整个核心基因组的0.01%)。人源KpII-B分离株09A323是2019年从希腊分离的,而其他两个牛源KpII-B(SD130-19和SD52-19)是2019年从中国分离的。尽管人源KpII-B分离株和两个牛源KpII-B分离株的核苷酸序列具有较高同源性,这表明KpII-B可能在人和牛之间传播,但尚不清楚这种传播是如何发生的。
最后,检测了来自奶牛和人类的KpII-B分离株的毒力因子,结果显示与大肠杆菌素(clbA-Q)和二元调控系统(kvgAS)合成相关的基因在人源(100%)和牛源(100%)KpII-B型肺炎克雷伯菌中都很常见,但很少检测到其他已知的毒力因子。奶牛源KpII-B分离株中尿囊素利用基因(allABCDRS、ylbEF、glc、fdrA和ybb等)的检出率(37.8%)远高于人源KpII-B分离株(13.9%)。人源(44.4%)和牛源(29.7%)的KpII-B肺炎克雷伯菌中,nif固氮操纵子相关基因的检出率很高,表明最初它们可能来源于植物[17,40] [附录A中的图 S2(b)]。
《4、 讨论》
4、 讨论
在本研究中对中国北方地区患CM奶牛中分离的肺炎克雷伯菌进行了系统性的分析,主要包括肺炎克雷伯菌流行率、抗生素敏感性、分子特征、种群结构,以及奶牛源与人源肺炎克雷伯菌分离株之间的关系。本研究报道的CM牛奶样品中肺炎克雷伯菌年平均流行率(2018年为3.0%,2019年为2.8%)与He等[25]之前报道的中国7个省份克雷伯菌属细菌的平均流行率(2.3%)基本一致,但低于中国东北牛群中克雷伯属细菌的平均流行率(14.4%, 183/1271)[41]。我们首次报道中国奶牛CM肺炎克雷伯菌分为三个种群,即KpI、KpII-B和KpIII,其中,KpI为最主要的种群,这些种群之前仅在美国的CM奶牛中发现[14],但他们的研究并没有对不同种群分离株的分子特征进行分析。本研究发现奶牛中KpI流行率高,而KpII-B和KpIII流行率低,但三个种群的分离株都呈现显著遗传多样性,这与其他关于人源肺炎克雷伯菌的研究结果类似[17,42]。
抗生素治疗是目前预防和治疗奶牛乳房炎的最有效手段。然而,抗生素的过度使用可能会引起泛耐药菌株的出现,导致奶牛乳房炎相关病原菌的抗生素耐药性增加[43]。在本研究中,KpI分离株对卡那霉素、阿莫西林/克拉维酸盐和头孢噻呋的耐药率分别为1.4%、1.4%和18.9%,低于Cheng等[21] 2019年对我国大型奶牛群的研究(分别为15%、38%和21%)。AmpC型β-内酰胺酶基因blaDHA和ESBL基因(blaCTX-M-14、blaCTX-M-15、blaSHV-27和blaSHV-2a)通常与人源KpI分离株的多药耐药性有关[44],在本研究中奶牛源KpI分离株中ESBL基因检出率较高(24.5%),但未检测到blaDHA基因。多项研究表明奶牛源肠杆菌科细菌中存在blaCTX-M-14和blaCTX-M-15 [14,22,45],但检出率较低,本研究结果与此基本一致;但本研究发现blaSHV-2a在KpI分离株中的检出率相对较高(15.4%),这并未在此前奶牛相关研究中报道过。本研究中的三个种群分离株的毒力基因携带情况调查显示,KpII-B型肺炎克雷伯菌中的铁摄取操纵子kfuABC和尿囊素利用基因(allABCDRS、ylbEF、glc、fdrA和ybb等)比KpI分离株中的丰富,而气杆菌素(iucA-D和iutA)、耶尔森菌素(irp、ybt和fyu)和二元调控系统基因(kvgAS)仅在KpI菌株中检测到,这些结果都与人类临床肺炎克雷伯菌分离株的研究结果一致。值得注意的是,I型菌毛调节基因fimK仅在KpI型肺炎克雷伯菌中被发现,有报道称它能增强肺炎克雷伯菌在小鼠肺炎模型中的致病性[46]。就耐药基因和毒力因子的携带情况而言,KpI分离株对奶牛的危害大于KpII-B和KpIII分离株。
KpI菌株是奶牛乳房炎的重要诱因,也是造成人类感染的常见病原菌[47]。然而,本研究显示,奶牛源和人源KpI分离株的种群结构(基因型)存在显著差异。ST11和ST23是人医临床分离株的主要克隆型,其中ST11主要与多药有关[48‒49],而ST23主要与高致病性有关[50‒51]。相比之下,在患CM的奶牛中,ST2324、ST107、ST116、ST43、ST111和ST2777型菌株更常见。与铁载体合成和荚膜调节相关的基因在人源KpI分离株中检出率高,在奶牛KpI分离株中罕见;但kfuABC在牛源KpI中明显比在人源KpI分离株中更普遍。种群结构和毒力基因携带的差异表明,奶牛源KpI对人类健康的威胁相对较小。
与奶牛源和人源KpI菌株在种群结构和潜在毒力因子携带方面存在显著差异相比,不同来源的KpII-B分离株更保守。例如,牛源和人源KpII-B分离株在进化树上共享分支,携带的已知毒力基因都很少。植物(如玉米)为克雷伯菌提供了合适的栖息地,克雷伯菌则能够产生固氮酶,有利于植物生长[40]。nif基因在所有KpII-B菌株中普遍存在,表明它们可能最初是植物共生细菌。此外,两个患CM奶牛分离的KpII-B菌株与人医临床KpII-B菌株具有高度的核苷酸序列一致性,表明KpII-B存在物种间传播的可能性。
此外,还发现奶牛KpI分离株中的clpC、lpfA、lac操纵子相关基因和Fe3+转运蛋白相关基因比人源KpI分离株丰富。这些基因可能对奶牛肺炎克雷伯菌的致病性或宿主适应性/特异性很重要。据报道,热激蛋白(HSP)ClpC是一种由clpC编码的ClpATPase,它会影响金黄色葡萄球菌在非专业吞噬细胞中的存活能力[52]。lpfA编码的LpfA是长极性菌毛(LPF)的主要纤维亚基结构,据报道LPF是牛乳房炎致病性大肠杆菌(MPEC)的关键毒力因子,与奶牛乳房炎形成过程中的上皮细胞侵袭有关[53‒55]。据报道,Fe3+转运蛋白相关基因(fecABDEIR)和lac乳糖操纵子可能有利于肺炎克雷伯菌利用宿主营养,从而赋予KpI选择性生长优势和适应性[14,17]。
《5、 结论》
5、 结论
在我国北方地区患临床型乳房炎的奶牛中,肺炎克雷伯菌的流行率较低。奶牛源肺炎克雷伯菌分离菌株包含三个种群(KpI、KpII-B和KpIII),其中,因KpI菌株的耐药基因和毒力基因的检测率比KpII-B和KpIII菌株更高,其对奶牛的危害性更大。奶牛源KpI分离株由于种群结构和携带的毒力基因存在较大差异,其对人类健康的威胁相对较小。kfuABC、clpC、lpfA、lac操纵子和Fe3+转运蛋白相关基因在奶牛源KpI分离株中检出率高,但在人源KpI分离株中很少检测到,表明这些基因可能是介导肺炎克雷伯菌感染奶牛乳腺的潜在毒力因子。此外,本研究的结果还表明,KpII-B分离株可能来源于植物,而且其在人与牛之间存在宿主间传播的可能,应予以监测。















 京公网安备 11010502051620号
京公网安备 11010502051620号




