《1、 引言》
1、 引言
抗生素生产过程是环境中抗生素污染的重要来源之一[1‒2]。抗生素选择压力可以促进环境中耐药基因和耐药细菌的增殖,进而加速环境耐药性发展,造成潜在的公共健康风险[3]。除废水外,发酵类抗生素生产过程还产生大量发酵菌渣。菌渣主要由生产菌株菌丝体、发酵培养基及残留的抗生素组成[4]。由于含有丰富的有机质,菌渣可以被资源化作为土壤有机肥或改良剂;然而,残留的抗生素限制了其资源化利用。对抗生素行业而言,将菌渣进行无害化处理后作为有机肥料施用,土壤具有良好的环境与经济效益。
红霉素是一种治疗革兰氏阳性菌感染的人畜共用的大环内酯类抗生素[5]。红霉素在环境中较稳定,在美国市政污水污泥中检出浓度最高达到(62.8 ± 5.8) μg∙kg-1 [6]。此外,由于对生态系统健康存在潜在影响,红霉素已被纳入欧盟水框架指令(Water Framework Directive of the European Union)的观察名录[7]。尽管使用和排放导致的真实地表水体中红霉素浓度高于效应水平的情形并不多见,但其生物风险仍不可忽视。欧盟水框架指令的观察名录实际上并没有将红霉素促进耐药性发展的风险考虑在内,然而耐药性选择可能比其他风险更有可能发生;由于红霉素对细菌的最小选择浓度(MIC)可以低至16 μg∙L-1 [8],因此预测的水环境最小无效应浓度(PNEC)仅为1 μg∙L-1。此外,将效应水平由水相向固相/半固相介质推演仍然是一个挑战[3]。
由于这些潜在的风险,红霉素发酵菌渣(erythromycin fermentation residue, EFR)是一个探究菌渣工业规模处理及后续土壤施用有效性与安全性的良好对象。据报道,水热处理可以将许多种发酵类抗生素(即由微生物发酵生产的抗生素)从水中去除,因为这类抗生素都有易水解的特性[9]。强化水解预处理的方法已经被用来去除污水中的抗生素,进而避免抗生素生产废水的生物处理系统中的耐药性发展[10‒12]。强化水解具有低成本和易工程化的优点[13],在实验室模拟实验中也被证实具有去除EFR中的红霉素的潜力[14]。然而,抗生素生产厂家使用水热处理的方法处理菌渣这类半固相废弃物仍然面临很多挑战。因此,探索将工业规模水热处理的菌渣用作土壤有机肥料是菌渣资源化研究的优先事项。
处理后的EFR降低了红霉素残留,进而可能减弱了对土壤微生物的选择压力。一项盆栽实验表明,处理后的红霉素菌渣施用导致土壤中含有300 μg∙kg-1的红霉素,但红霉素相关的耐药基因(如ermB、ermC和ermX)在结束实验的第49天时并未升高[15]。类似地,在一项处理后EFR的单次施用实验中也没有观察到土壤耐药基因的显著变化[16]。与此不同的是,一项向土壤中直接投加红霉素(10 mg∙kg-1)的多年连续暴露实验发现,红霉素的降解速率逐年升高,说明红霉素降解菌的富集[17];红霉素相关的耐药基因(只检测了msrE和mphE)在第4年暴露时开始升高[18]。这些结果表明,长期的、反复暴露的红霉素可能对土壤耐药基因存在逐渐发生的、缓慢累积的影响。目前,对这一过程的微生物生态机制的理解还知之甚少。
使用了处理后EFR的土壤中红霉素的残留往往处在细菌的亚抑制浓度水平(ng∙kg-1到μg∙kg-1)[15‒16],这与人类活动导致的环境介质中红霉素的污染水平相当[19]。土壤中残留的红霉素能够抑制群落中敏感菌(即对红霉素没有耐药能力的细菌)的生长,促进耐药基因通过水平转移来传播[20],以及促进环境细菌耐药性的进化进程[21‒22]。此外,通过菌渣施用也有可能向土壤中引入耐药细菌。因此,为了更深入地评估处理后EFR施用对土壤细菌群落耐药性的影响,需要开展长期、连续的土壤施用实验,采集时间序列样品,系统了解耐药基因及其相关决定因素的变化。
本文尝试在真实环境中进行红霉素菌渣对土壤生态系统影响的研究。具体目标包括:①调查水热处理的EFR及施用了一定条件下处理后的EFR的土壤中的红霉素残留;②评估处理后EFR土壤施用的累积影响;③解析水平和垂直传播机制在土壤细菌群落耐药性发展中的贡献。为此,本研究分析了红霉素生产企业中经工业规模一定条件下水热处理后的EFR,将处理后的EFR作为有机肥料开展了连续三年的土壤施用,并与施用粪肥、化肥做了对比。工业规模处理及多年土壤施用的研究结果将有助于EFR的风险评估和相关政策制定。
《2、 材料与方法》
2、 材料与方法
《2.1 工业规模的EFR处理》
2.1 工业规模的EFR处理
EFR是红霉素发酵生产的副产品,主要由失活的菌丝体、发酵培养基、残留的红霉素及发酵代谢产物等组成。2018—2020年,本研究调查了位于中国新疆维吾尔自治区的红霉素生产企业,该企业建立了一处工业规模的EFR水热处理设施。该企业的EFR年产量达100 000 t。EFR工业规模的无害化处理工艺包括水热处理(设计参数:160 ℃,大于15 min)和喷雾干燥(设计参数:450 ℃,大于5 s)。尽管水热反应的设计温度为160 ℃,但由于2018—2019年处于调试阶段,水热反应的实际操作温度为130~170 ℃;2020开始在水热处理工艺中增加了一个保温罐,相当于延长了高温处理时长。在喷雾干燥阶段,450 ℃的温度至少维持5 s,但整个升温和降温过程会持续至少20 min。
对新鲜的EFR(即未经水热处理的EFR)和处理后的EFR样品开展了DNA提取,并使用琼脂糖凝胶电泳的方法观察其DNA,目的是确认处理后的EFR中是否还有微生物存活。为了进一步评估可能残留的细菌DNA,还尝试开展了16S rRNA基因的聚合酶链反应(PCR)。
《2.2 处理后EFR的三年土壤施用》
2.2 处理后EFR的三年土壤施用
在位于中国新疆维吾尔自治区的未受人为扰动的地块上,开展了连续三年(2018—2020年)的处理后EFR土壤施用实验。种植的作物是大豆,原因是抗生素生产企业使用大豆作为发酵培养基。施肥剂量设置了农业常规剂量和极端高剂量。具体的,2018年包含对照组(CK)、低剂量组(EFR_L,3750 kg∙hm-2处理后的EFR)和高剂量组(EFR_H,7500 kg∙hm-2处理后的EFR)(见附录A中的图S1)。2019年为了探究极端剂量的影响,将处理后的EFR的施肥剂量加倍(EFR_L,7500 kg∙hm-2处理后的EFR;EFR_H,15 000 kg∙hm-2处理后的EFR);还增加了鸡粪肥和化肥施用的地块(见附录A中的图S1),原因是有报道称它们也会影响土壤耐药基因[23]。2020年延续2019年的实验设计不变。考虑到原位实验的复杂性,本研究采用了土壤中实测的红霉素浓度而不是施用的EFR的剂量,作为选择压力水平,以确保数据的可比性。
采集土壤样品时设置三个重复,并在大豆生长的三个阶段,即施肥前、施肥后(施肥后约2周)、成熟期(施肥后约3个月)采样。每个土壤样品被分为两部分,一部分在低温条件下转运至实验室并保存在-20 °C条件下,供后续DNA提取和红霉素测定;另一部分风干并过2 mm筛,供后续理化性质测定。2018年和2019年施肥前总计27个样品(3时期×3处理×3重复),2019年施肥后和成熟期总计36样品(2时期×6处理×3重复),2020年总计54样品(3时期×6处理×3重复)。更多的细节见附录A中的S1部分。
《2.3 化学分析》
2.3 化学分析
总碳(TC)、总氮(TN)使用元素分析仪(Vario MAX cube, Elementar, Germany)测定。为了测定红霉素浓度,使用Oasis HLB(500 mg,6 mL; Waters, USA)进行固相萃取[24],使用高效液相色谱双极质谱(UPLC-MS/MS; Waters)开展测试。更多的信息见附录A。
《2.4 DNA提取和16S rRNA基因扩增子测序》
2.4 DNA提取和16S rRNA基因扩增子测序
使用FastDNA SPIN Kit(MP Biomedicals, USA)进行DNA提取,使用NanoDrop1000 spectrophotometer(ThermoFisher, USA)进行DNA浓度和纯度测定。使用引物515F(5′-GTGCCAGCMGCCGCGG-3′)和907R(5′- CCGTCAATTCMTTTRAGTTT-3′)进行细菌16S rRNA基因V4~V5区的PCR扩增[25]。使用Illumina NovaSeq PE250进行高通量测序,委托广东美格基因科技有限公司开展实验。测序原始数据存储在美国国家生物信息技术中心(NCBI)生物项目管理数据库中(检索号:PRJNA650302)。更多细节见附录A中的S1部分。
《2.5 宏基因组测序定量耐药基因和可移动遗传元件(MGE)丰度》
2.5 宏基因组测序定量耐药基因和可移动遗传元件(MGE)丰度
使用Illumina X-ten进行宏基因组测序,委托广东美格基因科技有限公司开展实验。每个样品获得至少12 Gb测序数据。测序原始数据存储在NCBI生物项目管理数据库中(检索号:PRJNA647129)。质控后的测序数据[26]作为ARGs-OAP v2.2的输入数据,ARGs-OAP是从宏基因组数据中定量耐药基因的流程[27]。耐药基因的相对丰度以“拷贝数/细胞”来表示[27],这是一种宏基因组数据标准化的标准做法[28];细胞数用CopyRighter数据库来获得[29‒30]。基于NCBI和PlasmidFinder数据库建立了一个公共可移动遗传元件数据库[31],本研究基于该数据库,使用Salmon软件[32]获得了每个样品的可移动遗传元件的丰度信息。关于公共可移动遗传元件数据库的更多信息见附录A中的S1部分。
《2.6 宏基因组组装(assembly)和分箱(binning)》
2.6 宏基因组组装(assembly)和分箱(binning)
质控后的2020年施肥后和成熟期的测序数据使用MEGAHIT开展宏基因组组装[33]。每个样品都进行单独的组装,然后所有样品进行混合组装。宏基因组分箱使用metaWRAP流程[34],其中MaxBin2模块用于获得bin文件[35],Quant_bins模块用于计算每个bin在每个样品中的丰度。对高质量的基因组bin(基因组bin质量=完整度-5×污染度,> 50%)[36],使用基因组分类数据库(GTDB)-Tk软件确定其物种分类[37],使用综合性抗性研究数据库(Comprehensive Antibiotic Research Database, CARD)的耐药基因鉴别器(Resistance Gene Identifier, RGI)方法[38]和Prokka软件[39]确定其携带的耐药基因和可移动遗传元件。分箱得到的bin是基于宏基因组数据获得的重组的细菌基因组,提供了一种推测耐药基因的方法[40];这比基于相关性的推测有更高的可信度[41]。值得注意的是,分箱的方法在确定具有高移动性基因(如许多耐药基因)的宿主方面也不是完全可靠的。
《2.7 统计分析》
2.7 统计分析
非参数均值比较方法使用Mann-Whitney U检验或Kruskal-Wallis one-way analysis of variance(ANOVA)。不同样品之间的耐药基因、可移动遗传元件、细菌群落的Bray-Curtis相似度使用主坐标分析(PCoA)和置换多元方差分析来计算[42]。线性判别LEfSe分析[43]用于确定2020年实验组(处理后EFR施肥的样品)中被富集的耐药基因亚类。对于相关性分析,耐药基因和可移动遗传元件之间的关联性使用Spearman方法来计算,耐药基因与细菌群落的关联性使用Mantel检验和Procrustes检验来计算[44‒45]。结构方程模型(SEM)用于定量红霉素浓度、细菌群落、可移动遗传元件对耐药基因的直接和间接影响,结构方程模型的运算使用AMOS 22软件,并使用极大似然法[46]。模型需要满足一些拟合优度标准,包括不显著的χ2检验(P > 0.05)、拟合优度指数(GFI)大于0.90、均方根误差(RMSEA)小于0.08等[47]。
《3、 结果》
3、 结果
《3.1 工业化处理的EFR和施肥土壤中的红霉素浓度》
3.1 工业化处理的EFR和施肥土壤中的红霉素浓度
工业规模的水热处理和喷雾干燥能够减少EFR中的红霉素和水分含量,处理后的EFR用作土壤有机肥,更多的细节见附录A中的S2部分。2018—2020年,新鲜(未处理)的红霉素菌渣中红霉素含量为(1659 ± 202) mg∙kg-1干重,处理后的EFR中红霉素含量降低至(350 ± 12) mg∙kg-1干重(2018年)、(320 ± 26) mg∙kg-1干重(2019年)、(38 ± 1) mg∙kg-1干重(2020年)。2020年红霉素的去除率达到97.7%,原因是2020年增加了热储罐,相当于延长了水热处理时长。在琼脂糖凝胶电泳确认处理后的EFR中无法提取DNA(见附录A中的图S2),16S rRNA基因的PCR扩增也是失败的。因此,这一处理过程将EFR中的DNA降至不可检出的水平。
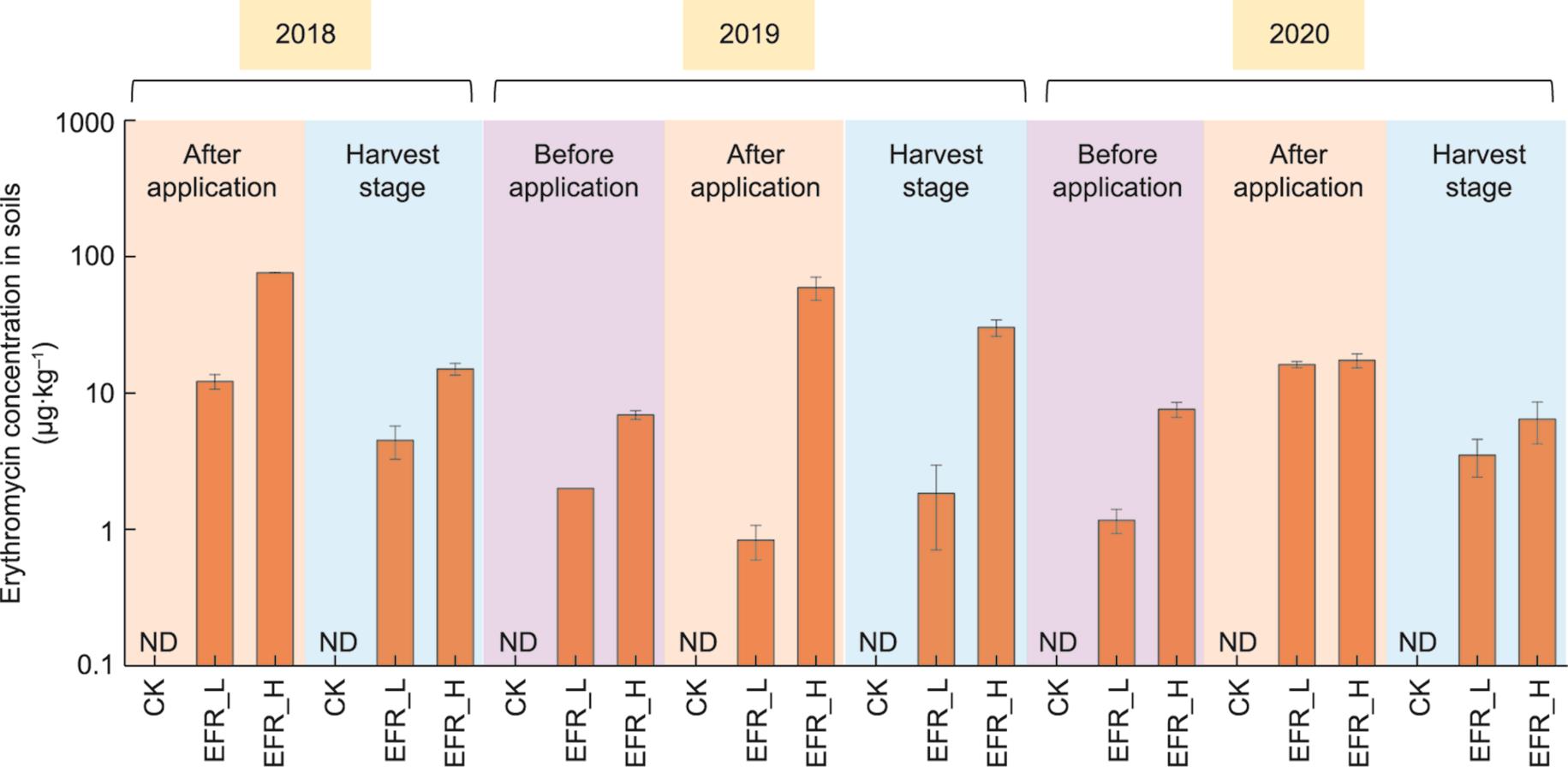
2018—2020年,处理后EFR作为有机肥土壤施用进行,同时开展粪肥和化肥施用作为对比(见附录A中的图S1)。相比于粪肥和化肥的施肥土壤,施用三年处理后的EFR的土壤中的总氮含量更高,但总碳含量类似(见附录A中的表S1和S2)。施用处理后的EFR的土壤中的红霉素含量为0.83~76.00 μg∙kg-1(见图1和附录A中的表S3)。整体而言,低剂量施肥土壤中红霉素含量低于高剂量施肥土壤;三年期间土壤中红霉素含量处于波动状态,没有明显的累积趋势;2019年和2020年施肥前土壤中红霉素含量仍在μg∙kg-1水平。对照土壤和施用粪肥、化肥的土壤中未检出红霉素(检出限为0.1 μg∙kg-1)(见附录A中的表S3)。
《图1》
图1 三年(2018年、2019年和2020年)期间施用处理后的EFR的土壤中的红霉素浓度。ND:未检出;EFR_L:2018年施用3750 kg·hm-2处理后的EFR的土壤或2019年、2020年施用7500 kg·hm-2处理后的EFR的土壤;EFR_H:2018年施用7500 kg·hm-2处理后的EFR的土壤或2019年、2020年施用15 000 kg·hm-2处理后的EFR的土壤。
《3.2 处理后的EFR三年施用导致耐药基因逐渐变化》
3.2 处理后的EFR三年施用导致耐药基因逐渐变化
基于宏基因组测序,施用处理后的EFR的土壤中总耐药基因的丰度在2018年和2019年与对照组没有显著差异(0.103~0.123拷贝/细胞;P > 0.05, Kruskal-Wallis one-way ANOVA)。然而,在2020年施肥后的阶段,施用处理后的EFR的剂量为7500 kg∙hm-2 [(0.187 ± 0.051)拷贝/细胞]和15 000 kg∙hm-2 [(0.278 ± 0.123)拷贝/细胞]的土壤中耐药基因的丰度显著高于对照组[(0.101 ± 0.003)拷贝/细胞] [见图2(a)及附录A中的表S4;P < 0.01, Kruskal-Wallis one-way ANOVA]。尽管粪肥和化肥施用也会增加土壤耐药基因丰度,但其幅度小于处理后的EFR的施用[见图2(a)和附录A中的表S4]。
《图2》
图2 施用处理后的EFR(treated EFR)、鸡粪肥(MF)、化肥(CF)的土壤中耐药基因丰度。施用实验持续三年(2018年、2019年、2020年),每年采集三个时期(施肥前、施肥后、成熟期)的样品。(a)宏基因组测序测定的土壤耐药基因总丰度,每个时期内标注相同字母的样品不具有统计学上的显著差异(基于Kruskal-Wallis one-way ANOVA)。(b)2020年三个时期的土壤耐药基因亚类,只展示组间有显著差异的基因亚类(基于LEfSe分析)。MF_L:施用7500 kg·hm-2鸡粪肥的土壤;MF_H:施用15 000 kg·hm-2鸡粪肥的土壤;CF:施用化肥的土壤。
LEfSe分析确定了2020年土壤中相比于对照组显著富集的耐药基因亚类,包括7个大环内酯类耐药基因[ erm(39)、ermB、ermF、ermT、macB、mgtA和oleD ]和20个其他类别的耐药基因。ermT、ermB和macB是丰度最高的(见附录A中的表S5~S7)。在2020年施肥后的阶段,总耐药基因和大环内酯类耐药基因的富集都是显著的,其中极端高剂量施肥土壤中大环内酯类耐药基因相比于对照组的变化倍数为12.59倍,而总耐药基因的变化倍数为2.75倍。这说明大环内酯类耐药基因被选择性地富集。
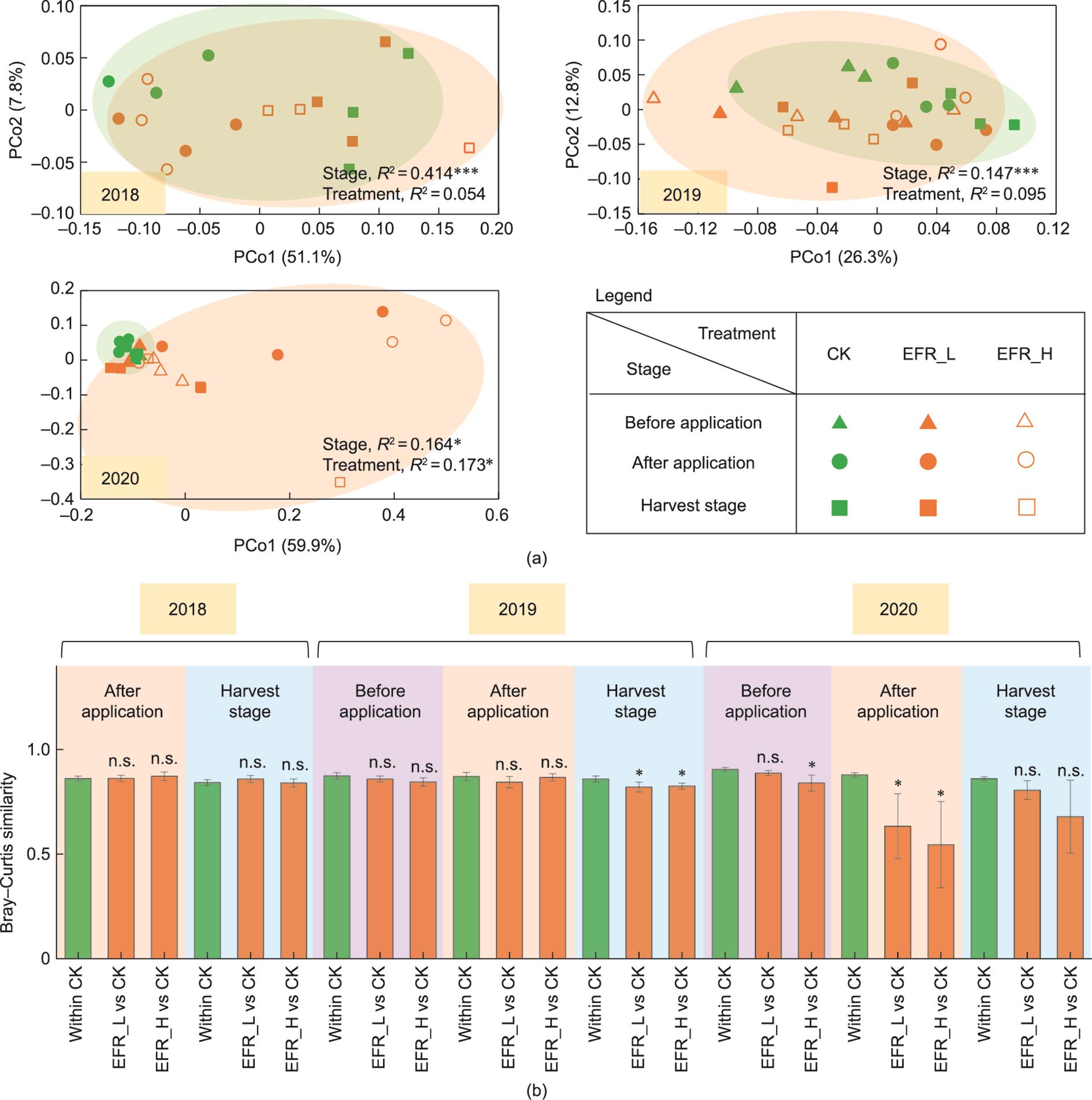
三年期间耐药基因多样性的变化趋势与丰度是类似的。主坐标分析和置换多元方差分析的结果表明,在2018年和2019年,三个阶段之间耐药基因β多样性的差异是显著的(P < 0.01, PERMANOVA),但处理后EFR施用的影响是微弱的(P > 0.05, PERMANOVA)。然而,到2020年,实验组与对照组的差异显著[图2(a);P < 0.05, PERMANOVA]。此外,还计算了每年每个阶段的实验组与对照组耐药基因的Bray-Curtis相似度,发现2019年成熟期、2020年施肥前、2020年施肥后的差异是显著的[图3(b);P < 0.05, Mann-Whitney U test]。
《图3》
图3 施用处理后的EFR的土壤中耐药基因多样性。施用实验持续三年(2018年、2019年、2020年),每年采集三个时期(施肥前、施肥后、成熟期)的样品。(a)耐药基因的主坐标分析(PCoA)和置换多元方差分析(PERMANOVA)。(b)实验组和对照组之间耐药基因的Bray-Curtis相似度。n.s.、*、**和***分别代表P ≥ 0.05、P < 0.05、P < 0.01和P < 0.001(基于Mann-Whitney U检验)。
总之,在三年实验期间,处理后的EFR被用作有机肥施用土壤,但其对土壤耐药基因的影响并没有立即显现。在施肥的第二年和第三年,耐药基因丰度和多样性的差异才逐渐出现。
《3.3 三年期间细菌群落和可移动遗传元件的动态》
3.3 三年期间细菌群落和可移动遗传元件的动态
基于宏基因组测序分析了三年期间细菌群落和可移动遗传元件的丰度和多样性。在门水平,放线菌门、拟杆菌门、厚壁菌门和变形菌门是主要的类别(见附录A中的图S3及表S8~S10)。细菌群落在2018年仅受施肥阶段的影响,而在2019年、2020年同时受到施肥阶段和处理后EFR施用的影响[见附录A中的图S4(a)]。Bray-Curtis相似度从2018年成熟期开始就与对照组显著不同。这些结果表明,细菌群落的差异比耐药基因的差异出现得更早。
可移动遗传元件(包括整合子、转座子、质粒、插入序列等)与耐药基因的水平转移密切相关。在2018年和2019年,实验组和对照组的可移动遗传元件的丰度相当(丰度为0.003~0.009拷贝/细胞;P > 0.05, Kruskal-Wallis one-way ANOVA)。然而,在2020年,实验组的可移动遗传元件丰度显著高于对照组。在极端高剂量处理后的EFR施用的土壤中检出最高丰度的可移动遗传元件(0.042拷贝/细胞)[见附录A中的图S5(a)和表S11]。丰度最高的可移动遗传元件为tnpA,其次是IS91和intI1 [见附录A中的图S5(b)和表S12~S14]。与耐药基因多样性类似,可移动遗传元件多样性在2018年和2019年仅受施肥阶段的影响,但在2020年开始受处理后EFR施用的影响(见附录A中的图S6)。
《3.4 可移动遗传元件、细菌群落、红霉素残留对耐药基因差异的贡献》
3.4 可移动遗传元件、细菌群落、红霉素残留对耐药基因差异的贡献
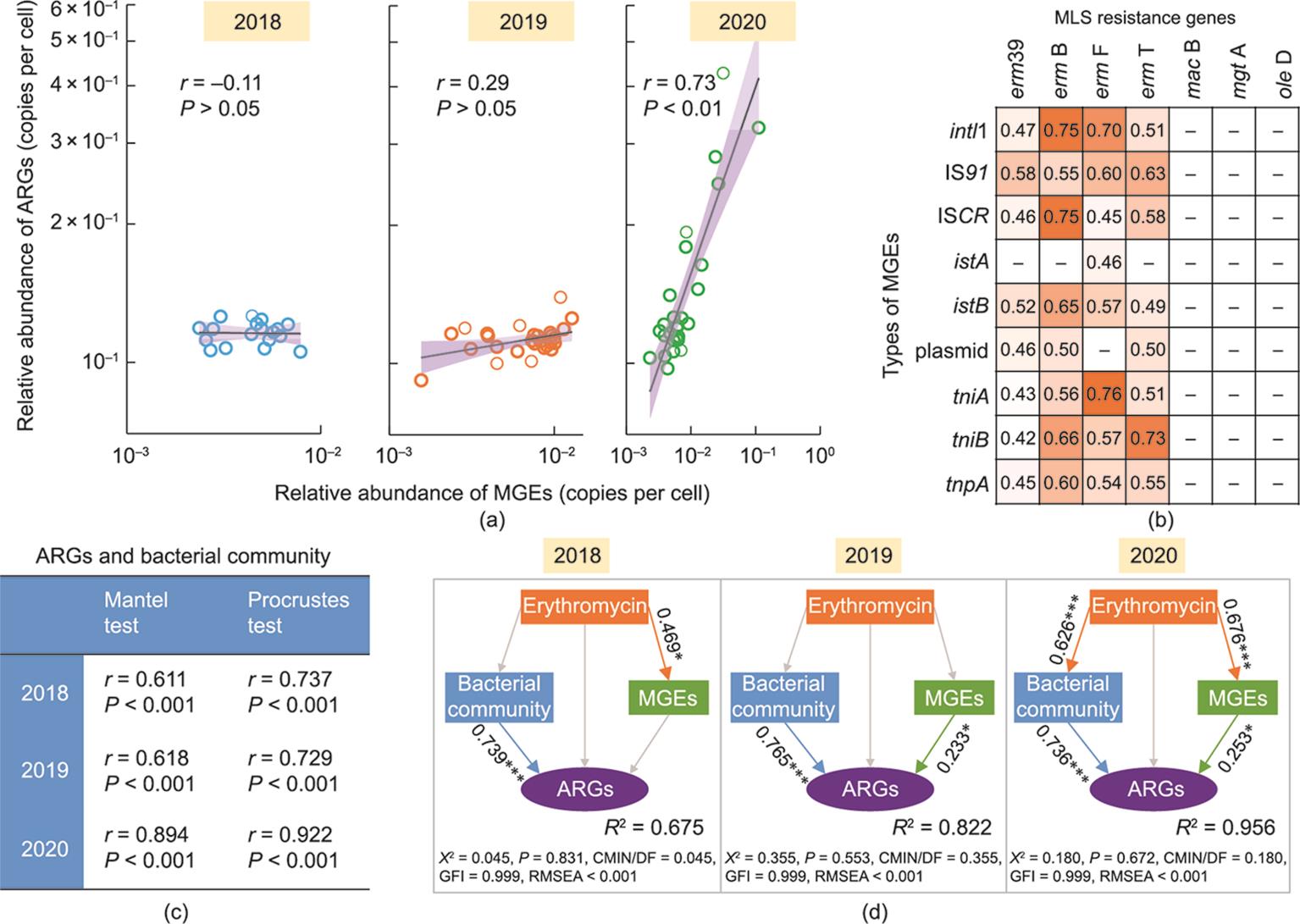
耐药基因与可移动遗传元件的相关性在2018年(Spearman’s r = -0.11, P > 0.05)和2019年(Spearman’s r = 0.29, P > 0.05)是不显著的,但在2020年是显著的(Spearman’s r = 0.73, P < 0.01)[图4(a)]。部分大环内酯类耐药基因[erm(39)、ermB、ermF和ermT ]与各类可移动遗传元件有普遍的相关性(P < 0.05,Spearman相关);然而,macB、mgtA和oleD与可移动遗传元件的相关性不显著[图4(b)]。细菌群落与耐药基因的相关性使用Mantel检验和Procrustes检验来计算。三年内的结果都是显著的,相关系数从2018年至2020年逐渐升高[图4(c)]。
《图4》
图4 三年实验期间耐药基因与可移动遗传元件、细菌群落、红霉素浓度之间的关联性。(a)三年实验期间土壤耐药基因相对丰度与可移动遗传元件相对丰度之间的Spearman相关性。(b)2020年土壤大环内酯类耐药基因相对丰度与可移动遗传元件大类相对丰度之间的Spearman相关性,图中仅展示统计学检验显著(P < 0.05)的结果。(c)三年实验期间土壤耐药基因与细菌群落的Mantel检验与Procrustes检验。(d)结构方程模型定量三年实验期间土壤红霉素浓度、细菌群落、可移动遗传元件对耐药基因的贡献。
为了更全面地理解可移动遗传元件、细菌群落、红霉素残留对耐药基因的影响,针对上述变量构建了结构方程模型。2020年,红霉素通过影响细菌群落和可移动遗传元件进而间接地影响耐药基因;耐药基因在2018年仅受细菌群落的影响,而在2019年和2020年同时受到细菌群落和可移动遗传元件的影响[图4(d)]。整体而言,在三年连续施肥的过程中,可移动遗传元件、细菌群落和红霉素对耐药基因的影响逐渐增强,对耐药基因差异的解释率由2018年的67.5%上升到2020年的95.6% [图4(d)]。
《3.5 宏基因组分箱推测耐药基因宿主的富集》
3.5 宏基因组分箱推测耐药基因宿主的富集
通过宏基因组组装和分箱可以分别得到携带耐药基因的contig和推测的耐药基因宿主,进而可以了解耐药基因的遗传环境与宿主丰度变化。在2020年施肥后和成熟期样品的组装结果中,得到68个携带耐药基因的contig,其中10个携带大环内酯类基因(ermB、ermT、macA和macB)。代表性的携带大环内酯类基因的contig如图5(a)所示。一种典型的可移动遗传元件,即Tn3家族转座酶基因与大环内酯类耐药基因erm共发生;大环内酯类耐药基因macA和macB与三磷酸腺苷结合盒转运蛋白(ABC transporter)基因共发生[图5(a)]。值得注意的是,通过对对照组样品进行组装没有得到携带大环内酯类基因的contig,说明只有高丰度的基因才有可能通过宏基因组组装得到其遗传环境信息。
《图5》
图5 (a)代表性的携带耐药基因的宏基因组组装的contig。(b)由宏基因组分箱得到的细菌基因组及其物种分类(基于GTDB)、相对丰度、携带耐药基因情况,仅展示高质量基因组(基因组bin质量=完整度‒5×污染度,>50%)。
通过宏基因组分箱获得了12个高质量的bin(基因组bin质量=完整度-5×污染度,> 50%),并使用GTDB-Tk软件确定了它们的物种组成。这些bin被注释为疣微菌门、变形菌门、拟杆菌门和放线菌门[见图5(b)及附录A中的表S15、表S16]。其中,9个bin携带大环内酯类耐药基因(macA、macB或oleD),这些bin在实验组的相对丰度高于对照组;与之相反,不携带大环内酯类耐药基因的bin(如bin.479和bin.62)的相对丰度在实验组和对照组之间相当,或者实验组低于对照组[见图5(b)及附录A中的表S15]。此外,携带大环内酯类基因的bin在实验组土壤中的富集也导致其他共存的耐药基因(如tetA)的富集[图2(b)和图5(b)]。
《4、 讨论》
4、 讨论
EFR的水热处理灭活了微生物并降解了DNA,因此,残余抗生素是菌渣肥料化利用过程中的主要风险。什么水平的抗生素残留量能够促进土壤耐药基因的富集与传播成为EFR安全处置与环境管理中的关键问题。本文首次报道了使用工业规模处理的EFR开展连续多年土壤施用的实验。一定条件下处理后的EFR的持续输入导致了土壤中相对稳定的亚抑制水平的红霉素残留。借助这样的实验设计可以探索处理后EFR施用对土壤的长期影响,并理解亚抑制抗生素导致的耐药性发展的内在机制。
《4.1 三年期间处理后EFR对土壤耐药基因的累积影响》
4.1 三年期间处理后EFR对土壤耐药基因的累积影响
土壤中红霉素浓度在施肥后升高,然后随时间延长又有所降低(图1)。环境中红霉素的转化过程包括吸附、水解、微生物降解和光解等[48]。因此,表面径流、渗透、水解和生物降解可能导致了红霉素的耗散[17]。然而,红霉素的耗散速率是有限的,三年期间红霉素残留保持在μg∙kg-1的水平[19]。土壤耐药基因及其决定因素(可移动遗传元件和细菌群落)也通过宏基因组测序来定量。这些生物变量对红霉素的响应都需要一定的时间才能显现。
细菌群落结构在第一年的成熟期就已经发生了变化,但耐药基因和可移动遗传元件分别到第二年和第三年才开始发生变化(见图2、图3和附录A中的表S3~S6)。值得注意的是,尽管2018年和2019年处理后的EFR中的红霉素浓度高于2020年处理后的EFR [图1(b)],但2018年和2019年土壤中的耐药基因丰度并没有显著升高(图2)。耐药基因的这种变化说明了长期亚抑制暴露的累积影响,这是无法通过单次暴露实验观察到的。此外,对于由粪肥施用引起的土壤耐药基因的富集没有观察到累积效应,可能的原因是粪肥施用引入的粪源细菌在土壤中难以长期定殖存活[49]。本研究中,大环内酯类耐药基因的富集程度高于其他耐药基因,直接说明红霉素的选择性压力的影响强于养分引入的非选择性影响。这种对土壤等环境介质的长期选择压力可能导致尚未发现的大环内酯类耐药性机制的进化(如富集与转移),最终可能造成临床的风险[3,50]。
《4.2 亚抑制红霉素压力下耐药基因富集的可能机制》
4.2 亚抑制红霉素压力下耐药基因富集的可能机制
为了解析亚抑制红霉素下耐药基因富集的机制,本研究系统利用了宏基因组测序、生物信息学分析和多元统计手段。前人的研究注意到长期红霉素暴露对msrE和mphE基因有影响[17‒18],本研究试图全面分析对各类耐药基因的影响,并解释水平和垂直传播的贡献。
在富集的大环内酯类耐药基因中,ermB、ermF和ermT是最占优势的[见图2(b)及附录A中的表S4~S7]。这些erm基因与多种可移动遗传元件有显著的相关性[图4(b)],并与Tn3家族转座酶基因共发生[图5(a)],但没有在由分箱得到的bin中发现erm基因[图5(b)]。综合上述结果,并考虑宏基因组分箱往往不能得到质粒信息[41],推测erm基因可能由质粒携带,并在红霉素的选择压力下通过水平转移的途径传播。水平转移在erm基因传播中的关键作用也被很多文献报道。大多数erm基因都是在质粒上被首次发现的;例如,ermT在质粒pGT633 [51‒52]和p121BS [53]上被发现,其遗传环境中存在IS1216V [54]。最近,红霉素被发现能够显著提高细菌接合转移效率[55],这进一步证实了本实验中可移动遗传元件的富集和质粒介导的水平转移的增强。
外排泵基因macA和macB能够介导细菌对14元环(如红霉素)和15元环大环内酯类药物的耐药[56‒57]。与erm基因不同的是,本研究中基于相关性分析[图4(b)]和宏基因组组装[图5(a)]发现,macA、macB与可移动遗传元件的关联性很弱,还发现macA、macB常出现在由宏基因组分箱得到的bin上[图5(b)]。上述结果表明,macA、macB倾向于位于细菌的染色体上,并在红霉素压力下通过垂直传播途径富集。
某些非大环内酯类基因也被富集[图2(b)],可能的原因是它们与大环内酯类基因的“共选择”。例如,tetA与macA、macB均在bin.594上,这个bin的富集意味着mac基因和tetA基因都被富集[图5(b)]。根据多元统计的结果,在三年实验期间,细菌群落(垂直途径)和可移动遗传元件(水平途径)对耐药基因变化的贡献都逐渐增强[图4(a)、(c)],结构方程模型的结果进一步印证这一趋势[图4(d)]。
由于亚抑制浓度的红霉素往往不会杀死敏感菌,但能够降低其生长速率[20,58‒59],因此亚抑制浓度的红霉素能够使耐药菌获得竞争优势,进而使耐药基因富集。此外,亚抑制浓度抗生素会增强细菌的SOS应答和RpoS调节,这些变化都能够增强耐药基因的水平转移[20]。
总之,在水平转移和垂直传播的共同作用下,三年期间使用处理后的EFR的土壤中耐药基因升高。在2020年成熟期,耐药基因丰度比施肥后低一些,可能的原因是质粒的适应性代价对细菌的增殖与存活产生负面影响[60]。不同的土著微生物群落和不同的土壤类型可能意味着对抗生素响应的不同[61]。未来的研究应该关注土壤类型的影响。
《4.3 环境与工程应用》
4.3 环境与工程应用
土壤施肥往往是连续多年的,因此,多年的监测和时间序列的采样对评估施肥影响至关重要。本研究中,红霉素降解速率比许多其他类型的抗生素要慢一些[9],连续三年施用形成了“引入-耗散”(entrance-dissipation)的动态变化。土壤中μg∙kg-1水平的红霉素残留持续了三年,耐药基因多样性与丰度分别在第二年和第三年开始发生变化。本研究强调,施用频率是评估土壤改良剂的环境影响时必须考虑的因素。此外,其他类型的发酵菌渣、粪肥[62]、污泥[63]和再生水[64]等都可能残留一定的抗生素,且都常被引入土壤中。基于本研究的结果,建议对上述可能向土壤引入抗生素的介质开展谨慎的评估。受野外试验的条件限制,本研究没有足够多的重复地块,未来的研究需要更严格的随机区组设计[65]。
在农业实践中,每年通过施用有机肥向土壤中引入的氮不宜超过250 kg∙hm-2 [66],处理后的EFR的氮含量约50 g∙kg-1。因此,本研究中的高剂量是一种极端剂量(每年向土壤中引入氮375~750 kg),而农业常规剂量(本实验中的低剂量)造成的耐药性发展是较弱的。施用剂量是处理后的EFR风险评估的关键问题。更长年限的农业常规剂量施肥是否会造成更高的耐药性风险,以及间隔年份施肥是否能够减弱耐药性风险,仍待进一步研究。
将抗生素发酵菌渣用作土壤改良剂有助于循环经济与碳中和[67],但必须谨慎对待可能带来的环境风险。热处理能够灭活微生物及其基因,因此有效去除菌渣中的残余抗生素成为菌渣安全资源化的关键。本研究中,工业规模的水热处理被用于去除EFR中的红霉素;水热处理还能够增强蛋白质类聚合物的释放,这使得菌渣能够产生更好的肥效或更好的生物能源转化潜力[14]。延长水热处理时间能够提高红霉素的去除率。此外,一些新技术包括催化强化的水热处理[68]和水热炭化[69]等,也是有潜力的菌渣无害化技术。
《5、 结论》
5、 结论
菌渣是由发酵类抗生素生产的副产物,是一种重要但常被忽视的环境中抗生素的污染源。本研究开展了连续多年的工业规模的EFR处理与土壤大田施用实验。EFR中红霉素的去除率可达到97.7%,三年连续施用向土壤中引入μg∙kg-1水平的红霉素残留。极端高剂量处理后的EFR的土壤施用对耐药基因产生了累积影响,但农业常规剂量施用的影响较弱。此外,宏基因组数据的系统挖掘表明,erm基因和mac基因富集分别通过水平途径和垂直途径实现。这些发现对抗生素菌渣及其他含抗生素的固废的风险评估与环境管理将会有所帮助。















 京公网安备 11010502051620号
京公网安备 11010502051620号




